
| Cancer | Chr | Position | Mutation Type | dbSNP | Protein-change | Allele Freq | RBD |
|---|---|---|---|---|---|---|---|
| ACC | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CHOL | |||||||
| CHOL | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| ESCA | |||||||
| ESCA | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| LAML | |||||||
| LGG | |||||||
| LGG | |||||||
| LGG | |||||||
| LGG | |||||||
| LGG | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| PAAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| SARC | |||||||
| SARC | |||||||
| SARC | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| TGCT | |||||||
| TGCT | |||||||
| THCA | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCS | |||||||
| UCS | |||||||
| UCS |
| Cancer | Type | Freq | Q-value |
|---|---|---|---|
| BRCA | |||
| DLBC | |||
| HNSC | |||
| LUSC | |||
| OV | |||
| PAAD | |||
| READ | |||
| TGCT | |||
| THYM |
| Cancer | P-value | Q-value |
|---|---|---|
| MESO | ||
| ACC | ||
| SKCM | ||
| ESCA | ||
| KIRP | ||
| BLCA | ||
| LIHC | ||
| LGG | ||
| CHOL | ||
| UVM | ||
| OV |
| Transcript ID | Name | Length | RefSeq ID | Protein ID | Length | RefSeq ID | UniportKB ID |
|---|---|---|---|---|---|---|---|
| ENST00000545285 | WNK1-219 | 528 | - | ENSP00000446462 | 176 (aa) | - | H0YH79 |
| ENST00000447667 | WNK1-203 | 1902 | - | ENSP00000392542 | 393 (aa) | - | F6UYG0 |
| ENST00000544965 | WNK1-217 | 653 | - | ENSP00000438313 | 218 (aa) | - | H0YFF1 |
| ENST00000544559 | WNK1-216 | 226 | - | ENSP00000437503 | 76 (aa) | - | H0YF81 |
| ENST00000545055 | WNK1-218 | 624 | - | - | - (aa) | - | - |
| ENST00000574679 | WNK1-221 | 722 | - | ENSP00000459380 | 240 (aa) | - | I3L251 |
| ENST00000340908 | WNK1-202 | 11208 | XM_011521001 | ENSP00000341292 | 2634 (aa) | XP_011519303 | Q9H4A3 |
| ENST00000574564 | WNK1-220 | 1401 | - | ENSP00000460651 | 434 (aa) | - | A0A0B4J299 |
| ENST00000540360 | WNK1-211 | 668 | - | - | - (aa) | - | - |
| ENST00000537687 | WNK1-209 | 11232 | XM_011520997 | ENSP00000444465 | 2642 (aa) | XP_011519299 | Q9H4A3 |
| ENST00000540885 | WNK1-212 | 874 | - | - | - (aa) | - | - |
| ENST00000542543 | WNK1-214 | 506 | - | - | - (aa) | - | - |
| ENST00000538787 | WNK1-210 | 579 | - | - | - (aa) | - | - |
| ENST00000535698 | WNK1-207 | 736 | - | ENSP00000439552 | 245 (aa) | - | H0YFP5 |
| ENST00000535572 | WNK1-206 | 9886 | XM_011521008 | ENSP00000441972 | 2134 (aa) | XP_011519310 | F5GWT4 |
| ENST00000537603 | WNK1-208 | 553 | - | - | - (aa) | - | - |
| ENST00000542424 | WNK1-213 | 912 | - | - | - (aa) | - | - |
| ENST00000315939 | WNK1-201 | 10452 | XM_006719003 | ENSP00000313059 | 2382 (aa) | XP_006719066 | Q9H4A3 |
| ENST00000543065 | WNK1-215 | 201 | - | ENSP00000438985 | 67 (aa) | - | H0YFK5 |
| ENST00000534872 | WNK1-205 | 584 | - | ENSP00000446253 | 194 (aa) | - | H0YH68 |
| ENST00000530271 | WNK1-204 | 11804 | - | ENSP00000433548 | 2833 (aa) | - | Q9H4A3 |
| ensgID | Trait | pValue | Pubmed ID |
|---|---|---|---|
| ENSG00000060237 | Blood Pressure | 1.7526904E-005 | - |
| ENSG00000060237 | Blood Pressure | 7.5949174E-005 | - |
| ENSG00000060237 | Blood Pressure | 7.7059493E-005 | - |
| ENSG00000060237 | Body Mass Index | 6E-6 | 25673413 |
| ENSG00000060237 | Body Mass Index | 2E-7 | 25673413 |
| ENSG00000060237 | Body Mass Index | 2E-7 | 25673413 |
| ENSG00000060237 | Colorectal Neoplasms | 3E-6 | 24836286 |
| ensgID | SNP | Chromosome | Position | SNP-risk | Trait | PubmedID | 95% CI | Or or BEAT | EFO ID |
|---|---|---|---|---|---|---|---|---|---|
| ENSG00000060237 | rs7953330 | 12 | 889653 | G | Lung cancer | 28604730 | [1.06-1.12] | 1.0911875 | EFO_0001071 |
| ENSG00000060237 | rs7953330 | 12 | 889653 | G | Squamous cell lung carcinoma | 28604730 | [1.11-1.2] | 1.1567484 | EFO_0000708 |
| ENSG00000060237 | rs4619206 | 12 | 893691 | C | Lung cancer in ever smokers | 28604730 | [1.07-1.14] | 1.1038703 | EFO_0006527|EFO_0001071 |
| ENSG00000060237 | rs12301299 | 12 | 882544 | C | Blood protein levels | 29875488 | [0.19-0.31] unit increase | 0.25 | EFO_0007937 |
| ENSG00000060237 | rs12309274 | 12 | 866782 | T | Colorectal cancer | 24836286 | [1.06-1.16] | 1.11 | EFO_0005842 |
| ENSG00000060237 | rs11613704 | 12 | 871759 | A | Eosinophil percentage of white cells | 27863252 | [0.021-0.038] unit increase | 0.02968211 | EFO_0007991 |
| ENSG00000060237 | rs11613704 | 12 | 871759 | A | Eosinophil counts | 27863252 | [0.021-0.038] unit increase | 0.02963015 | EFO_0004842 |
| ENSG00000060237 | rs11611246 | 12 | 830314 | T | Body mass index | 26426971 | unit increase | 0.024242502 | EFO_0004340 |
| ENSG00000060237 | rs11611246 | 12 | 830314 | G | Body mass index | 30108127 | [NR] unit decrease | 0.022 | EFO_0004340 |
| ENSG00000060237 | rs372936466 | 12 | 864111 | ? | Heel bone mineral density | 30048462 | [0.017-0.034] unit increase | 0.0253698 | EFO_0009270 |
| ENSG00000060237 | rs55726687 | 12 | 882140 | ? | Body mass index | 30595370 | EFO_0004340 | ||
| ENSG00000060237 | rs10774463 | 12 | 792399 | ? | Red cell distribution width | 30595370 | EFO_0005192 |
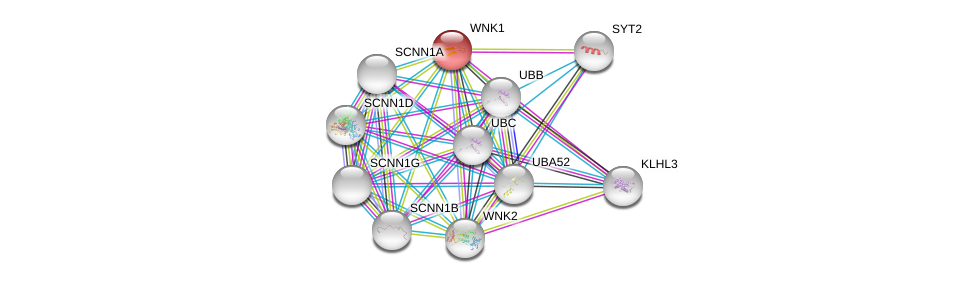
| Go ID | Go_term | PubmedID | Evidence | Category |
|---|---|---|---|---|
| GO:0000287 | magnesium ion binding | - | IEA | Function |
| GO:0002028 | regulation of sodium ion transport | - | ISS | Process |
| GO:0003084 | positive regulation of systemic arterial blood pressure | - | IEA | Process |
| GO:0004672 | protein kinase activity | 16669787. | IMP | Function |
| GO:0004674 | protein serine/threonine kinase activity | 21873635. | IBA | Function |
| GO:0004674 | protein serine/threonine kinase activity | 10660600. | IDA | Function |
| GO:0004674 | protein serine/threonine kinase activity | 27400149. | ISS | Function |
| GO:0004674 | protein serine/threonine kinase activity | 24393035. | TAS | Function |
| GO:0005515 | protein binding | 15350218.16832045.17721439.19389623. | IPI | Function |
| GO:0005524 | ATP binding | 10660600. | IDA | Function |
| GO:0005737 | cytoplasm | 21873635. | IBA | Component |
| GO:0005737 | cytoplasm | 10660600. | IDA | Component |
| GO:0005829 | cytosol | 21873635. | IBA | Component |
| GO:0005829 | cytosol | - | IDA | Component |
| GO:0005829 | cytosol | - | ISS | Component |
| GO:0006468 | protein phosphorylation | 21873635. | IBA | Process |
| GO:0006468 | protein phosphorylation | 10660600. | IDA | Process |
| GO:0006468 | protein phosphorylation | 16669787. | IMP | Process |
| GO:0006811 | ion transport | - | ISS | Process |
| GO:0010766 | negative regulation of sodium ion transport | 21873635. | IBA | Process |
| GO:0010820 | positive regulation of T cell chemotaxis | 27400149. | IMP | Process |
| GO:0010923 | negative regulation of phosphatase activity | 19389623. | IDA | Process |
| GO:0016020 | membrane | - | ISS | Component |
| GO:0018105 | peptidyl-serine phosphorylation | 27400149. | ISS | Process |
| GO:0018107 | peptidyl-threonine phosphorylation | 24393035. | TAS | Process |
| GO:0019869 | chloride channel inhibitor activity | 21873635. | IBA | Function |
| GO:0019869 | chloride channel inhibitor activity | 17673510. | IDA | Function |
| GO:0019870 | potassium channel inhibitor activity | 21873635. | IBA | Function |
| GO:0019901 | protein kinase binding | 16669787. | IPI | Function |
| GO:0019902 | phosphatase binding | 19389623. | IDA | Function |
| GO:0023016 | signal transduction by trans-phosphorylation | 16669787. | IDA | Process |
| GO:0030291 | protein serine/threonine kinase inhibitor activity | - | IEA | Function |
| GO:0030295 | protein kinase activator activity | 16669787. | IMP | Function |
| GO:0032147 | activation of protein kinase activity | - | IEA | Process |
| GO:0032414 | positive regulation of ion transmembrane transporter activity | 21873635. | IBA | Process |
| GO:0033633 | negative regulation of cell-cell adhesion mediated by integrin | 27400149. | IMP | Process |
| GO:0034115 | negative regulation of heterotypic cell-cell adhesion | 27400149. | IMP | Process |
| GO:0034260 | negative regulation of GTPase activity | 27400149. | IMP | Process |
| GO:0035556 | intracellular signal transduction | 21873635. | IBA | Process |
| GO:0035556 | intracellular signal transduction | 10660600. | IDA | Process |
| GO:0035556 | intracellular signal transduction | 24393035. | TAS | Process |
| GO:0038116 | chemokine (C-C motif) ligand 21 signaling pathway | 27400149. | ISS | Process |
| GO:0046777 | protein autophosphorylation | - | IEA | Process |
| GO:0048666 | neuron development | 15060842. | NAS | Process |
| GO:0050794 | regulation of cellular process | - | ISS | Process |
| GO:0050801 | ion homeostasis | 21873635. | IBA | Process |
| GO:0050852 | T cell receptor signaling pathway | 27400149. | ISS | Process |
| GO:0071277 | cellular response to calcium ion | - | IEA | Process |
| GO:0071901 | negative regulation of protein serine/threonine kinase activity | - | IEA | Process |
| GO:0090188 | negative regulation of pancreatic juice secretion | - | IEA | Process |
| GO:0090263 | positive regulation of canonical Wnt signaling pathway | 23797875. | IMP | Process |
| GO:0097022 | lymphocyte migration into lymph node | 27400149. | ISS | Process |
| GO:1903038 | negative regulation of leukocyte cell-cell adhesion | 27400149. | IMP | Process |
| GO:1903288 | positive regulation of potassium ion import | 21873635. | IBA | Process |
| GO:1990869 | cellular response to chemokine | 27400149. | IMP | Process |
| GO:2000651 | positive regulation of sodium ion transmembrane transporter activity | 21873635. | IBA | Process |