
| Cancer | Chr | Position | Mutation Type | dbSNP | Protein-change | Allele Freq | RBD |
|---|---|---|---|---|---|---|---|
| ACC | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| CESC | |||||||
| CESC | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| HNSC | |||||||
| KIRC | |||||||
| LGG | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUSC | |||||||
| LUSC | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| PRAD | |||||||
| READ | |||||||
| READ | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| THCA | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC |
| Cancer | Type | Freq | Q-value |
|---|---|---|---|
| ACC | |||
| BRCA | |||
| CHOL | |||
| COAD | |||
| LGG | |||
| LIHC | |||
| LUAD | |||
| PAAD | |||
| READ | |||
| TGCT | |||
| THYM | |||
| UCS |
| Cancer | P-value | Q-value |
|---|---|---|
| KIRC | ||
| STAD | ||
| UCS | ||
| SKCM | ||
| TGCT | ||
| KIRP | ||
| BLCA | ||
| CESC | ||
| READ | ||
| LAML | ||
| LIHC | ||
| LGG | ||
| UVM |
| Transcript ID | Name | Length | RefSeq ID | Protein ID | Length | RefSeq ID | UniportKB ID |
|---|---|---|---|---|---|---|---|
| ENST00000368547 | ECHS1-201 | 1617 | - | ENSP00000357535 | 290 (aa) | - | P30084 |
| Pathway ID | Pathway Name | Source |
|---|---|---|
| hsa00062 | Fatty acid elongation | KEGG |
| hsa00071 | Fatty acid degradation | KEGG |
| hsa00280 | Valine, leucine and isoleucine degradation | KEGG |
| hsa00310 | Lysine degradation | KEGG |
| hsa00380 | Tryptophan metabolism | KEGG |
| hsa00410 | beta-Alanine metabolism | KEGG |
| hsa00640 | Propanoate metabolism | KEGG |
| hsa00650 | Butanoate metabolism | KEGG |
| hsa01100 | Metabolic pathways | KEGG |
| hsa01200 | Carbon metabolism | KEGG |
| hsa01212 | Fatty acid metabolism | KEGG |
| ensgID | Trait | pValue | Pubmed ID |
|---|---|---|---|
| ENSG00000127884 | Longevity | 3E-10 | 25918517 |
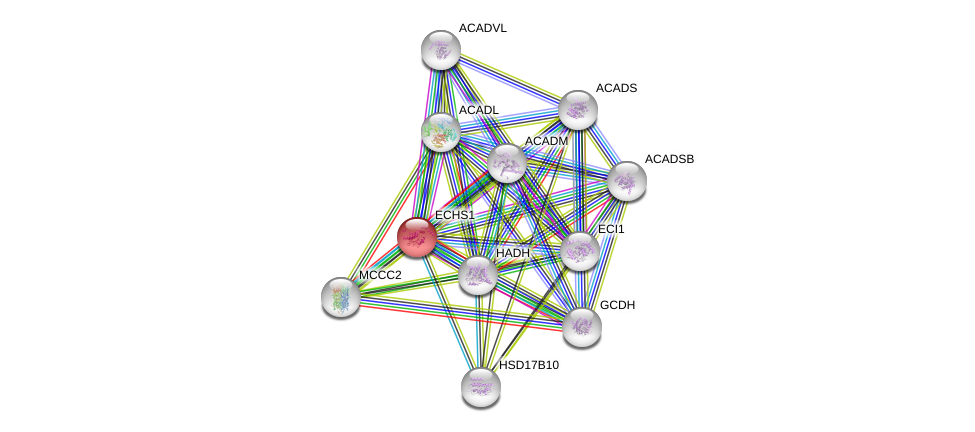
| Ensembl ID | Gene Symbol | Coverage | Identiy | Ortholog | Gene Symbol | Coverage | Identiy | Species |
|---|---|---|---|---|---|---|---|---|
| ENSG00000127884 | ECHS1 | 89 | 61.479 | WBGene00001155 | ech-6 | 89 | 61.479 | Caenorhabditis_elegans |
| ENSG00000127884 | ECHS1 | 95 | 57.091 | FBgn0033879 | CG6543 | 93 | 57.091 | Drosophila_melanogaster |
| ENSG00000127884 | ECHS1 | 100 | 87.586 | ENSMUSG00000025465 | Echs1 | 100 | 87.586 | Mus_musculus |
| Go ID | Go_term | PubmedID | Evidence | Category |
|---|---|---|---|---|
| GO:0004300 | enoyl-CoA hydratase activity | 21873635. | IBA | Function |
| GO:0004300 | enoyl-CoA hydratase activity | 26251176. | IDA | Function |
| GO:0005515 | protein binding | 14557246.23416296.24510904.24947832. | IPI | Function |
| GO:0005739 | mitochondrion | 21873635. | IBA | Component |
| GO:0005739 | mitochondrion | - | IDA | Component |
| GO:0005739 | mitochondrion | 16130169. | TAS | Component |
| GO:0005759 | mitochondrial matrix | - | TAS | Component |
| GO:0006635 | fatty acid beta-oxidation | 21873635. | IBA | Process |
| GO:0006635 | fatty acid beta-oxidation | 26251176. | IDA | Process |
| GO:0006635 | fatty acid beta-oxidation | - | IEA | Process |
| GO:0006635 | fatty acid beta-oxidation | - | TAS | Process |