
| Cancer | Chr | Position | Mutation Type | dbSNP | Protein-change | Allele Freq | RBD |
|---|---|---|---|---|---|---|---|
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| ESCA | |||||||
| ESCA | |||||||
| ESCA | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| LAML | |||||||
| LAML | |||||||
| LAML | |||||||
| LGG | |||||||
| LGG | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| PAAD | |||||||
| PAAD | |||||||
| PAAD | |||||||
| PAAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| SARC | |||||||
| SARC | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| THCA | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCS | |||||||
| UCS |
| Cancer | Type | Freq | Q-value |
|---|---|---|---|
| CESC | |||
| PAAD | |||
| PAAD |
| Cancer | P-value | Q-value |
|---|---|---|
| THYM | ||
| KIRC | ||
| SARC | ||
| MESO | ||
| HNSC | ||
| SKCM | ||
| LUSC | ||
| ESCA | ||
| KIRP | ||
| CESC | ||
| UCEC | ||
| LIHC | ||
| LGG |
| Transcript ID | Name | Length | RefSeq ID | Protein ID | Length | RefSeq ID | UniportKB ID |
|---|---|---|---|---|---|---|---|
| ENST00000451437 | MYO1B-209 | 1310 | - | ENSP00000388140 | 303 (aa) | - | E7EQD9 |
| ENST00000496992 | MYO1B-214 | 561 | - | - | - (aa) | - | - |
| ENST00000420448 | MYO1B-206 | 374 | - | ENSP00000387610 | 83 (aa) | - | C9K0I9 |
| ENST00000471904 | MYO1B-211 | 770 | - | - | - (aa) | - | - |
| ENST00000490069 | MYO1B-212 | 2111 | - | - | - (aa) | - | - |
| ENST00000461714 | MYO1B-210 | 562 | - | - | - (aa) | - | - |
| ENST00000392318 | MYO1B-204 | 5082 | XM_005246572 | ENSP00000376132 | 1136 (aa) | XP_005246629 | O43795 |
| ENST00000438652 | MYO1B-208 | 542 | - | ENSP00000399459 | 149 (aa) | - | C9JUP5 |
| ENST00000494129 | MYO1B-213 | 1880 | - | - | - (aa) | - | - |
| ENST00000427152 | MYO1B-207 | 791 | - | ENSP00000408454 | 192 (aa) | - | H7C2Y7 |
| ENST00000634375 | MYO1B-215 | 567 | - | ENSP00000489544 | 189 (aa) | - | A0A0U1RRI3 |
| ENST00000304164 | MYO1B-201 | 4933 | - | ENSP00000306382 | 1136 (aa) | - | O43795 |
| ENST00000392316 | MYO1B-203 | 4764 | XM_024452914 | ENSP00000376130 | 1107 (aa) | XP_024308682 | E9PDF6 |
| ENST00000339514 | MYO1B-202 | 5026 | XM_024452915 | ENSP00000341903 | 1078 (aa) | XP_024308683 | O43795 |
| ENST00000418908 | MYO1B-205 | 572 | - | ENSP00000401324 | 163 (aa) | - | C9JYW1 |
| ensgID | Trait | pValue | Pubmed ID |
|---|---|---|---|
| ENSG00000128641 | Body Weight | 2.54864114150903E-6 | 17903300 |
| ENSG00000128641 | Body Weight | 1.68783670470051E-8 | 17903300 |
| ENSG00000128641 | Body Mass Index | 4.50706366908064E-6 | 17903300 |
| ENSG00000128641 | gamma-Glutamyltransferase | 1E-11 | 22001757 |
| ENSG00000128641 | Energy Intake | 9E-6 | 23251661 |
| ENSG00000128641 | Antineoplastic Agents | 9E-6 | 23648065 |
| ENSG00000128641 | Fluorouracil | 9E-6 | 23648065 |
| ensgID | SNP | Chromosome | Position | SNP-risk | Trait | PubmedID | 95% CI | Or or BEAT | EFO ID |
|---|---|---|---|---|---|---|---|---|---|
| ENSG00000128641 | rs1823913 | 2 | 191253321 | A | Obesity-related traits | 23251661 | [NR] g/d increase | 0.03 | EFO_0003939 |
| ENSG00000128641 | rs13030978 | 2 | 191252512 | T | Liver enzyme levels (gamma-glutamyl transferase) | 22001757 | [2.80-4.6] % increase | 3.7 | EFO_0004532 |
| ENSG00000128641 | rs1947457 | 2 | 191298970 | ? | Adolescent idiopathic scoliosis | 30019117 | EFO_0005423 |
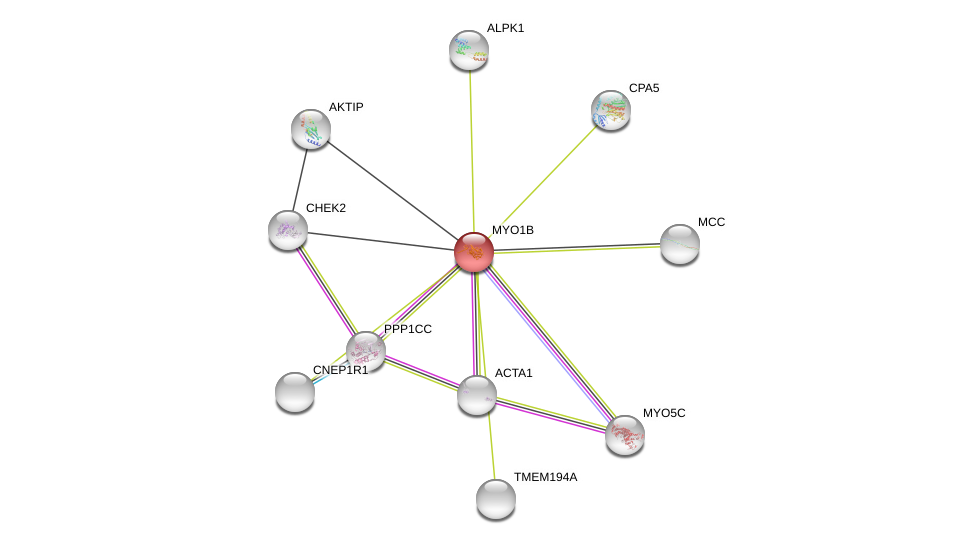
| Go ID | Go_term | PubmedID | Evidence | Category |
|---|---|---|---|---|
| GO:0000146 | microfilament motor activity | 21666684. | IMP | Function |
| GO:0000146 | microfilament motor activity | - | ISS | Function |
| GO:0005516 | calmodulin binding | - | IEA | Function |
| GO:0005524 | ATP binding | 21666684. | IMP | Function |
| GO:0005524 | ATP binding | - | ISS | Function |
| GO:0005546 | phosphatidylinositol-4,5-bisphosphate binding | - | ISS | Function |
| GO:0005547 | phosphatidylinositol-3,4,5-trisphosphate binding | - | ISS | Function |
| GO:0005737 | cytoplasm | - | ISS | Component |
| GO:0005769 | early endosome | 16219689. | IDA | Component |
| GO:0005884 | actin filament | 16219689. | IDA | Component |
| GO:0005886 | plasma membrane | - | IDA | Component |
| GO:0005886 | plasma membrane | - | ISS | Component |
| GO:0005903 | brush border | - | ISS | Component |
| GO:0006892 | post-Golgi vesicle-mediated transport | 21666684. | IMP | Process |
| GO:0007015 | actin filament organization | 21666684. | IMP | Process |
| GO:0010008 | endosome membrane | 16219689. | IDA | Component |
| GO:0016459 | myosin complex | - | IEA | Component |
| GO:0030048 | actin filament-based movement | - | ISS | Process |
| GO:0030175 | filopodium | - | ISS | Component |
| GO:0030898 | actin-dependent ATPase activity | - | IEA | Function |
| GO:0032588 | trans-Golgi network membrane | 21666684. | IDA | Component |
| GO:0045177 | apical part of cell | - | IEA | Component |
| GO:0045296 | cadherin binding | 25468996. | HDA | Function |
| GO:0048471 | perinuclear region of cytoplasm | 21666684. | IDA | Component |
| GO:0051015 | actin filament binding | 21666684. | IMP | Function |
| GO:0051017 | actin filament bundle assembly | - | ISS | Process |
| GO:0070062 | extracellular exosome | 19056867.19199708. | HDA | Component |
| GO:0071944 | cell periphery | - | ISS | Component |