
| Cancer | Chr | Position | Mutation Type | dbSNP | Protein-change | Allele Freq | RBD |
|---|---|---|---|---|---|---|---|
| ACC | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CHOL | |||||||
| CHOL | |||||||
| CHOL | |||||||
| CHOL | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| DLBC | |||||||
| ESCA | |||||||
| ESCA | |||||||
| ESCA | |||||||
| ESCA | |||||||
| ESCA | |||||||
| ESCA | |||||||
| ESCA | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| LAML | |||||||
| LAML | |||||||
| LGG | |||||||
| LGG | |||||||
| LGG | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| MESO | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| PAAD | |||||||
| PAAD | |||||||
| PAAD | |||||||
| PAAD | |||||||
| PAAD | |||||||
| PAAD | |||||||
| PCPG | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| SARC | |||||||
| SARC | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| TGCT | |||||||
| TGCT | |||||||
| THCA | |||||||
| THCA | |||||||
| THCA | |||||||
| THYM | |||||||
| THYM | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCS |
| Cancer | Type | Freq | Q-value |
|---|---|---|---|
| ACC | |||
| BLCA | |||
| BRCA | |||
| CESC | |||
| KIRC | |||
| KIRP | |||
| LGG | |||
| MESO | |||
| OV | |||
| PAAD | |||
| SKCM | |||
| THYM |
| Cancer | P-value | Q-value |
|---|---|---|
| THYM | ||
| KIRC | ||
| STAD | ||
| SARC | ||
| MESO | ||
| SKCM | ||
| PRAD | ||
| BRCA | ||
| CESC | ||
| READ | ||
| LAML | ||
| LGG | ||
| LUAD | ||
| OV |
| Transcript ID | Name | Length | RefSeq ID | Protein ID | Length | RefSeq ID | UniportKB ID |
|---|---|---|---|---|---|---|---|
| ENST00000366806 | AFDN-203 | 5962 | - | ENSP00000355771 | 1781 (aa) | - | A8MQ02 |
| ENST00000486649 | AFDN-214 | 423 | - | - | - (aa) | - | - |
| ENST00000366809 | AFDN-204 | 4839 | - | - | - (aa) | - | - |
| ENST00000511503 | AFDN-220 | 1257 | - | - | - (aa) | - | - |
| ENST00000497596 | AFDN-215 | 728 | - | ENSP00000426462 | 167 (aa) | - | H0YA98 |
| ENST00000392112 | AFDN-206 | 7459 | XM_006715494 | ENSP00000375960 | 1743 (aa) | XP_006715557 | P55196 |
| ENST00000485634 | AFDN-213 | 606 | - | ENSP00000423035 | 191 (aa) | - | H0Y948 |
| ENST00000447894 | AFDN-211 | 5475 | XM_005266996 | ENSP00000404595 | 1824 (aa) | XP_005267053 | P55196 |
| ENST00000351017 | AFDN-202 | 5823 | XM_006715491 | ENSP00000252692 | 1831 (aa) | XP_006715554 | J3KN01 |
| ENST00000515794 | AFDN-222 | 746 | - | ENSP00000422166 | 124 (aa) | - | H0Y8U8 |
| ENST00000400822 | AFDN-207 | 7593 | - | ENSP00000383623 | 1834 (aa) | - | P55196 |
| ENST00000509296 | AFDN-219 | 686 | - | - | - (aa) | - | - |
| ENST00000400824 | AFDN-208 | 875 | - | ENSP00000383625 | 247 (aa) | - | F8W9I4 |
| ENST00000511637 | AFDN-221 | 5999 | - | - | - (aa) | - | - |
| ENST00000400825 | AFDN-209 | 963 | - | ENSP00000383626 | 250 (aa) | - | E9PFY5 |
| ENST00000507679 | AFDN-217 | 670 | - | - | - (aa) | - | - |
| ENST00000476946 | AFDN-212 | 867 | - | ENSP00000424188 | 129 (aa) | - | H0Y9I0 |
| ENST00000392108 | AFDN-205 | 6812 | - | ENSP00000375956 | 1651 (aa) | - | P55196 |
| ENST00000507704 | AFDN-218 | 1031 | - | ENSP00000421441 | 313 (aa) | - | H0Y8L4 |
| ENST00000503021 | AFDN-216 | 581 | - | - | - (aa) | - | - |
| ENST00000423229 | AFDN-210 | 1807 | - | ENSP00000414675 | 389 (aa) | - | H0Y7R8 |
| ENST00000344191 | AFDN-201 | 5249 | - | ENSP00000341118 | 1665 (aa) | - | Q5TIG5 |
| ensgID | Trait | pValue | Pubmed ID |
|---|---|---|---|
| ENSG00000130396 | Fibrinogen | 1.0580000E-005 | - |
| ENSG00000130396 | Fibrinogen | 2.0590000E-007 | - |
| ENSG00000130396 | Platelet Function Tests | 7.2200000E-006 | - |
| ENSG00000130396 | Platelet Function Tests | 9.4600000E-006 | - |
| ENSG00000130396 | Platelet Function Tests | 5.8900000E-007 | - |
| ENSG00000130396 | Platelet Function Tests | 1.0500000E-006 | - |
| ENSG00000130396 | Platelet Function Tests | 6.1000000E-007 | - |
| ENSG00000130396 | Platelet Function Tests | 1.1700000E-007 | - |
| ENSG00000130396 | Platelet Function Tests | 3.9900000E-006 | - |
| ENSG00000130396 | Platelet Function Tests | 2.9000000E-006 | - |
| ENSG00000130396 | Platelet Function Tests | 1.4700000E-007 | - |
| ENSG00000130396 | Platelet Function Tests | 3.4600000E-008 | - |
| ENSG00000130396 | Platelet Function Tests | 3.5600000E-006 | - |
| ENSG00000130396 | Child Development Disorders, Pervasive | 6.1424800E-005 | - |
| ensgID | SNP | Chromosome | Position | SNP-risk | Trait | PubmedID | 95% CI | Or or BEAT | EFO ID |
|---|---|---|---|---|---|---|---|---|---|
| ENSG00000130396 | rs62427705 | 6 | 167886940 | ? | Non-albumin protein levels | 29403010 | [0.023-0.046] unit increase novel | 0.03439 | EFO_0004568 |
| ENSG00000130396 | rs3800533 | 6 | 167935400 | ? | Red cell distribution width | 30595370 | EFO_0005192 | ||
| ENSG00000130396 | rs2285248 | 6 | 167942931 | ? | Red blood cell count | 30595370 | EFO_0004305 |
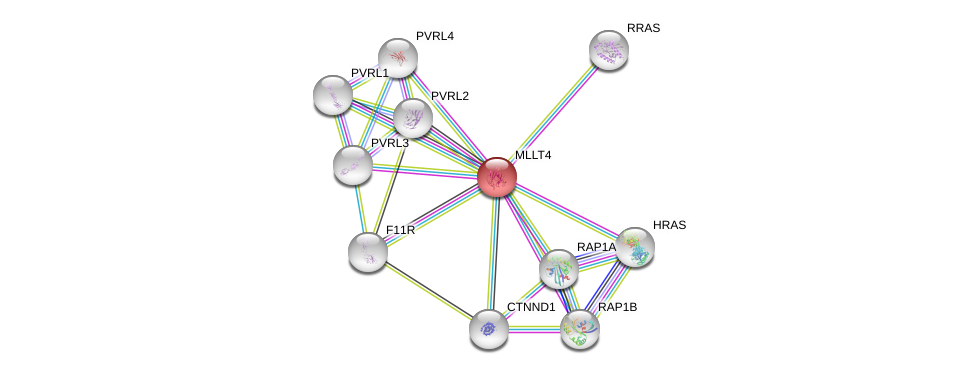
| Ensembl ID | Gene Symbol | Coverage | Identiy | Ortholog | Gene Symbol | Coverage | Identiy | Species |
|---|---|---|---|---|---|---|---|---|
| ENSG00000130396 | AFDN | 100 | 96.761 | ENSMUSG00000068036 | Afdn | 100 | 96.943 | Mus_musculus |
| Go ID | Go_term | PubmedID | Evidence | Category |
|---|---|---|---|---|
| GO:0005515 | protein binding | 17491594.22891260.23750010.23885123.27815408. | IPI | Function |
| GO:0005654 | nucleoplasm | - | IDA | Component |
| GO:0005829 | cytosol | - | IDA | Component |
| GO:0005829 | cytosol | - | TAS | Component |
| GO:0005886 | plasma membrane | - | IDA | Component |
| GO:0005911 | cell-cell junction | 10856295. | TAS | Component |
| GO:0005913 | cell-cell adherens junction | 21873635. | IBA | Component |
| GO:0007155 | cell adhesion | 10856295. | TAS | Process |
| GO:0007165 | signal transduction | - | IEA | Process |
| GO:0007267 | cell-cell signaling | 8242616. | TAS | Process |
| GO:0008022 | protein C-terminus binding | 10856295. | TAS | Function |
| GO:0010628 | positive regulation of gene expression | 25893857. | IMP | Process |
| GO:0016607 | nuclear speck | - | IDA | Component |
| GO:0017016 | Ras GTPase binding | 23885123. | IPI | Function |
| GO:0022409 | positive regulation of cell-cell adhesion | 25893857. | IMP | Process |
| GO:0030054 | cell junction | - | IDA | Component |
| GO:0030336 | negative regulation of cell migration | 21478912. | IMP | Process |
| GO:0032880 | regulation of protein localization | 21873635. | IBA | Process |
| GO:0034332 | adherens junction organization | - | TAS | Process |
| GO:0043547 | positive regulation of GTPase activity | 23885123. | IMP | Process |
| GO:0044291 | cell-cell contact zone | 16882694. | IDA | Component |
| GO:0044331 | cell-cell adhesion mediated by cadherin | 23990464. | ISS | Process |
| GO:0045296 | cadherin binding | 25468996. | HDA | Function |
| GO:0050839 | cell adhesion molecule binding | 21873635. | IBA | Function |
| GO:0051015 | actin filament binding | 16882694. | IDA | Function |
| GO:0061951 | establishment of protein localization to plasma membrane | 23990464. | ISS | Process |
| GO:0070160 | tight junction | 27815408. | IDA | Component |
| GO:0070830 | bicellular tight junction assembly | 27815408. | IMP | Process |
| GO:0090557 | establishment of endothelial intestinal barrier | 23885123. | IMP | Process |
| GO:2000049 | positive regulation of cell-cell adhesion mediated by cadherin | 16882694. | IMP | Process |