
| Cancer | Chr | Position | Mutation Type | dbSNP | Protein-change | Allele Freq | RBD |
|---|---|---|---|---|---|---|---|
| ACC | |||||||
| ACC | |||||||
| ACC | |||||||
| ACC | |||||||
| ACC | |||||||
| ACC | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CHOL | |||||||
| CHOL | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| ESCA | |||||||
| ESCA | |||||||
| ESCA | |||||||
| ESCA | |||||||
| ESCA | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| KICH | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| LAML | |||||||
| LAML | |||||||
| LGG | |||||||
| LGG | |||||||
| LGG | |||||||
| LGG | |||||||
| LGG | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| PAAD | |||||||
| PAAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| SARC | |||||||
| SARC | |||||||
| SARC | |||||||
| SARC | |||||||
| SARC | |||||||
| SARC | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| THCA | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCS | |||||||
| UVM | |||||||
| UVM |
| Cancer | Type | Freq | Q-value |
|---|---|---|---|
| LIHC | |||
| LUAD | |||
| MESO |
| Cancer | P-value | Q-value |
|---|---|---|
| THYM | ||
| KIRC | ||
| SARC | ||
| MESO | ||
| ACC | ||
| PRAD | ||
| KIRP | ||
| COAD | ||
| PAAD | ||
| PCPG | ||
| KICH | ||
| LIHC | ||
| CHOL | ||
| LUAD | ||
| UVM |
| Transcript ID | Name | Length | RefSeq ID | Protein ID | Length | RefSeq ID | UniportKB ID |
|---|---|---|---|---|---|---|---|
| ENST00000514974 | CENPE-206 | 581 | - | ENSP00000426023 | 183 (aa) | - | D6RHK2 |
| ENST00000611174 | CENPE-208 | 8260 | - | ENSP00000483542 | 2664 (aa) | - | A0A087X0P0 |
| ENST00000509823 | CENPE-205 | 515 | - | - | - (aa) | - | - |
| ENST00000515478 | CENPE-207 | 291 | - | - | - (aa) | - | - |
| ENST00000265148 | CENPE-201 | 8612 | XM_011531544 | ENSP00000265148 | 2701 (aa) | XP_011529846 | Q02224 |
| ENST00000503705 | CENPE-203 | 3468 | - | ENSP00000423981 | 1126 (aa) | - | D6RBW0 |
| ENST00000509120 | CENPE-204 | 792 | - | - | - (aa) | - | - |
| ENST00000380026 | CENPE-202 | 8241 | XM_011531547 | ENSP00000369365 | 2580 (aa) | XP_011529849 | Q02224 |
| ensgID | Trait | pValue | Pubmed ID |
|---|---|---|---|
| ENSG00000138778 | Stroke | 9.9178495E-004 | - |
| ENSG00000138778 | C-Reactive Protein | 8.945e-005 | 19060910 |
| ENSG00000138778 | C-Reactive Protein | 4.701e-005 | 19060910 |
| ENSG00000138778 | C-Reactive Protein | 9.209e-006 | 19060910 |
| ENSG00000138778 | Platelet Function Tests | 1.0807500E-005 | - |
| ENSG00000138778 | Platelet Function Tests | 1.8164200E-005 | - |
| ENSG00000138778 | Platelet Function Tests | 2.4002800E-005 | - |
| ENSG00000138778 | Sex Hormone-Binding Globulin | 3.4020000E-005 | - |
| ENSG00000138778 | Sex Hormone-Binding Globulin | 3.4690000E-005 | - |
| ENSG00000138778 | Sex Hormone-Binding Globulin | 3.8210000E-005 | - |
| ENSG00000138778 | Testicular Neoplasms | 2E-10 | 23666240 |
| ENSG00000138778 | Testicular Neoplasms | 2E-7 | 19483681 |
| ENSG00000138778 | Testicular Germ Cell Tumor | 6E-12 | 25877299 |
| ensgID | SNP | Chromosome | Position | SNP-risk | Trait | PubmedID | 95% CI | Or or BEAT | EFO ID |
|---|---|---|---|---|---|---|---|---|---|
| ENSG00000138778 | rs2720460 | 4 | 103133529 | A | Testicular germ cell tumor | 25877299 | [NR] | 1.32 | EFO_1000566 |
| ENSG00000138778 | rs2720460 | 4 | 103133529 | A | Testicular germ cell tumor | 23666240 | [1.12-1.36] | 1.24 | EFO_0005088 |
| ENSG00000138778 | rs2720460 | 4 | 103133529 | G | Testicular germ cell tumor | 28604732 | [1.20-1.35] | 1.2820514 | EFO_1000566 |
| ENSG00000138778 | rs13114629 | 4 | 103111211 | ? | General cognitive ability | 29844566 | z-score decrease | 4.903 | EFO_0004337 |
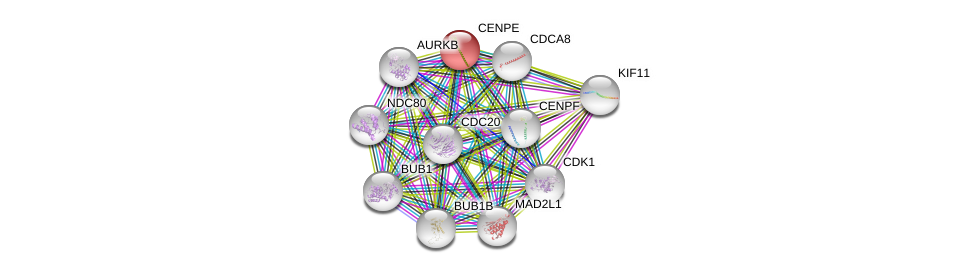
| Ensembl ID | Gene Symbol | Coverage | Identiy | Ortholog | Gene Symbol | Coverage | Identiy | Species |
|---|---|---|---|---|---|---|---|---|
| ENSG00000138778 | CENPE | 100 | 73.233 | ENSMUSG00000045328 | Cenpe | 100 | 69.884 | Mus_musculus |
| Go ID | Go_term | PubmedID | Evidence | Category |
|---|---|---|---|---|
| GO:0000278 | mitotic cell cycle | 24748105. | IMP | Process |
| GO:0000775 | chromosome, centromeric region | 2022189.11084331.26321640. | IDA | Component |
| GO:0000776 | kinetochore | 9763420.11682612.12925705.25395579.25918224. | IDA | Component |
| GO:0000777 | condensed chromosome kinetochore | - | IEA | Component |
| GO:0000779 | condensed chromosome, centromeric region | 19465021. | IDA | Component |
| GO:0003777 | microtubule motor activity | 21873635. | IBA | Function |
| GO:0003777 | microtubule motor activity | 25908662. | IDA | Function |
| GO:0003777 | microtubule motor activity | 7889940. | IMP | Function |
| GO:0005515 | protein binding | 9763420.17535814.25918224.26321640. | IPI | Function |
| GO:0005524 | ATP binding | - | IEA | Function |
| GO:0005634 | nucleus | 9763420. | IMP | Component |
| GO:0005654 | nucleoplasm | - | IDA | Component |
| GO:0005694 | chromosome | 2022189. | IDA | Component |
| GO:0005828 | kinetochore microtubule | 25908662. | IDA | Component |
| GO:0005829 | cytosol | - | IDA | Component |
| GO:0005829 | cytosol | - | TAS | Component |
| GO:0005871 | kinesin complex | 21873635. | IBA | Component |
| GO:0005874 | microtubule | 21873635. | IBA | Component |
| GO:0005874 | microtubule | 7889940. | IDA | Component |
| GO:0006890 | retrograde vesicle-mediated transport, Golgi to ER | - | TAS | Process |
| GO:0007018 | microtubule-based movement | 21873635. | IBA | Process |
| GO:0007018 | microtubule-based movement | - | TAS | Process |
| GO:0007052 | mitotic spindle organization | 25908662. | IMP | Process |
| GO:0007059 | chromosome segregation | 24748105. | IMP | Process |
| GO:0007079 | mitotic chromosome movement towards spindle pole | 25908662. | IDA | Process |
| GO:0007080 | mitotic metaphase plate congression | 21873635. | IBA | Process |
| GO:0007080 | mitotic metaphase plate congression | 15843429. | IMP | Process |
| GO:0007275 | multicellular organism development | - | IEA | Process |
| GO:0008017 | microtubule binding | 21873635. | IBA | Function |
| GO:0008017 | microtubule binding | 25908662. | IDA | Function |
| GO:0015630 | microtubule cytoskeleton | - | IDA | Component |
| GO:0016020 | membrane | 19946888. | HDA | Component |
| GO:0016887 | ATPase activity | 21873635. | IBA | Function |
| GO:0019886 | antigen processing and presentation of exogenous peptide antigen via MHC class II | - | TAS | Process |
| GO:0030071 | regulation of mitotic metaphase/anaphase transition | 2022189. | IMP | Process |
| GO:0030496 | midbody | 2022189.9763420. | IDA | Component |
| GO:0043515 | kinetochore binding | 9763420. | IDA | Function |
| GO:0045860 | positive regulation of protein kinase activity | 12925705. | IMP | Process |
| GO:0051233 | spindle midzone | 9763420. | IDA | Component |
| GO:0051301 | cell division | - | IEA | Process |
| GO:0051310 | metaphase plate congression | 23891108.25395579.25743205.25908662. | IMP | Process |
| GO:0051315 | attachment of mitotic spindle microtubules to kinetochore | 15843429. | IMP | Process |
| GO:0051382 | kinetochore assembly | 9763420. | IDA | Process |
| GO:0099606 | microtubule plus-end directed mitotic chromosome migration | 25908662. | IDA | Process |
| GO:0099607 | lateral attachment of mitotic spindle microtubules to kinetochore | 23891108. | IMP | Process |
| GO:1990023 | mitotic spindle midzone | 2022189. | IDA | Component |