
| Cancer | Chr | Position | Mutation Type | dbSNP | Protein-change | Allele Freq | RBD |
|---|---|---|---|---|---|---|---|
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| ESCA | |||||||
| ESCA | |||||||
| ESCA | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| LGG | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| PAAD | |||||||
| PAAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| READ | |||||||
| SARC | |||||||
| SARC | |||||||
| SARC | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| THCA | |||||||
| THYM | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC |
| Cancer | Type | Freq | Q-value |
|---|---|---|---|
| BLCA | |||
| DLBC | |||
| GBM | |||
| KIRC | |||
| LGG | |||
| PCPG | |||
| READ | |||
| SARC | |||
| TGCT | |||
| THYM | |||
| UCEC |
| Cancer | P-value | Q-value |
|---|---|---|
| KIRC | ||
| SARC | ||
| MESO | ||
| ACC | ||
| PCPG | ||
| LAML | ||
| KICH | ||
| UCEC | ||
| OV |
| Transcript ID | Name | Length | RefSeq ID | Protein ID | Length | RefSeq ID | UniportKB ID |
|---|---|---|---|---|---|---|---|
| ENST00000464656 | TP53BP2-204 | 593 | - | ENSP00000417987 | 64 (aa) | - | H0Y847 |
| ENST00000494100 | TP53BP2-212 | 701 | - | ENSP00000420225 | 234 (aa) | - | H7C5L8 |
| ENST00000473135 | TP53BP2-207 | 567 | - | - | - (aa) | - | - |
| ENST00000465119 | TP53BP2-205 | 465 | - | ENSP00000418252 | 47 (aa) | - | F8WBY9 |
| ENST00000489310 | TP53BP2-210 | 533 | - | - | - (aa) | - | - |
| ENST00000483398 | TP53BP2-209 | 1841 | - | ENSP00000417174 | 64 (aa) | - | H0Y847 |
| ENST00000343537 | TP53BP2-201 | 4651 | XM_011544268 | ENSP00000341957 | 1134 (aa) | XP_011542570 | Q13625 |
| ENST00000472180 | TP53BP2-206 | 467 | - | - | - (aa) | - | - |
| ENST00000391878 | TP53BP2-202 | 4741 | XM_011544269 | ENSP00000375750 | 1005 (aa) | XP_011542571 | Q13625 |
| ENST00000481128 | TP53BP2-208 | 631 | - | - | - (aa) | - | - |
| ENST00000464172 | TP53BP2-203 | 537 | - | - | - (aa) | - | - |
| ENST00000496282 | TP53BP2-213 | 481 | - | - | - (aa) | - | - |
| ENST00000498843 | TP53BP2-214 | 2781 | - | - | - (aa) | - | - |
| ENST00000490896 | TP53BP2-211 | 577 | - | - | - (aa) | - | - |
| Pathway ID | Pathway Name | Source |
|---|---|---|
| hsa04390 | Hippo signaling pathway | KEGG |
| ensgID | SNP | Chromosome | Position | SNP-risk | Trait | PubmedID | 95% CI | Or or BEAT | EFO ID |
|---|---|---|---|---|---|---|---|---|---|
| ENSG00000143514 | rs12723025 | 1 | 223789817 | ? | Triptolide cytotoxicity | 26121980 | unit increase | 0.56 | EFO_0007662|EFO_0006952 |
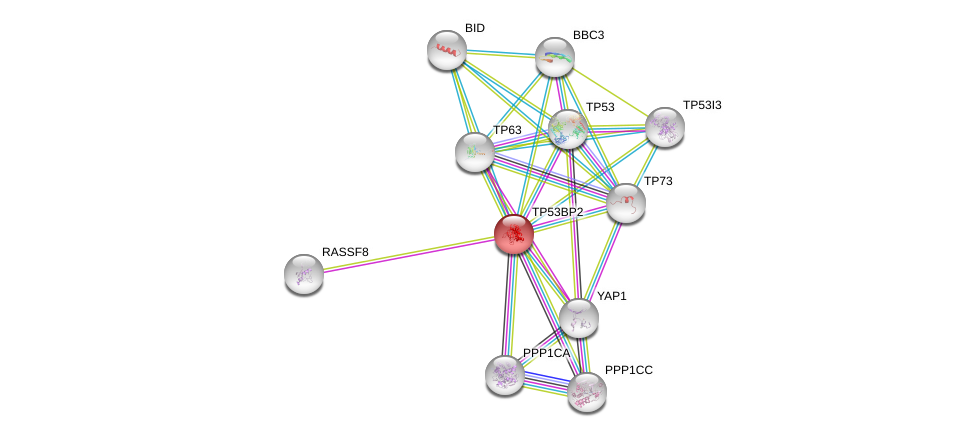
| Go ID | Go_term | PubmedID | Evidence | Category |
|---|---|---|---|---|
| GO:0002039 | p53 binding | 21873635. | IBA | Function |
| GO:0002039 | p53 binding | 14985081. | IPI | Function |
| GO:0005070 | SH3/SH2 adaptor activity | 8668206. | TAS | Function |
| GO:0005515 | protein binding | 8875926.10498867.10646860.11278849.11684014.12694406.14729977.14985081.15161933.15231748.18275817.18448430.18719108.19377511.21513714.21988832.21998301.22321011.24366813.24855949.25344754.25436413.26496610.28330616. | IPI | Function |
| GO:0005634 | nucleus | 21873635. | IBA | Component |
| GO:0005634 | nucleus | 19377511. | IDA | Component |
| GO:0005654 | nucleoplasm | - | TAS | Component |
| GO:0005737 | cytoplasm | 10646860.14985081.19377511. | IDA | Component |
| GO:0005737 | cytoplasm | 15782125. | TAS | Component |
| GO:0005739 | mitochondrion | - | IEA | Component |
| GO:0005829 | cytosol | - | IDA | Component |
| GO:0007049 | cell cycle | - | IEA | Process |
| GO:0007165 | signal transduction | 9748285. | TAS | Process |
| GO:0017124 | SH3 domain binding | - | IEA | Function |
| GO:0030054 | cell junction | - | IDA | Component |
| GO:0042802 | identical protein binding | 18448430. | IPI | Function |
| GO:0042981 | regulation of apoptotic process | - | TAS | Process |
| GO:0045786 | negative regulation of cell cycle | 14729977. | TAS | Process |
| GO:0048471 | perinuclear region of cytoplasm | 14985081. | IDA | Component |
| GO:0048471 | perinuclear region of cytoplasm | 10646860. | IDA | Component |
| GO:0051059 | NF-kappaB binding | 10498867. | IPI | Function |
| GO:0072332 | intrinsic apoptotic signaling pathway by p53 class mediator | 11684014. | IDA | Process |
| GO:1900119 | positive regulation of execution phase of apoptosis | 14985081. | IMP | Process |
| GO:1900740 | positive regulation of protein insertion into mitochondrial membrane involved in apoptotic signaling pathway | - | TAS | Process |
| GO:1901216 | positive regulation of neuron death | - | IEA | Process |
| GO:1901796 | regulation of signal transduction by p53 class mediator | - | TAS | Process |