
| Cancer | Chr | Position | Mutation Type | dbSNP | Protein-change | Allele Freq | RBD |
|---|---|---|---|---|---|---|---|
| ACC | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CHOL | |||||||
| CHOL | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| ESCA | |||||||
| ESCA | |||||||
| ESCA | |||||||
| ESCA | |||||||
| ESCA | |||||||
| ESCA | |||||||
| ESCA | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| KICH | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| LGG | |||||||
| LGG | |||||||
| LGG | |||||||
| LGG | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| PAAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| SARC | |||||||
| SARC | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| TGCT | |||||||
| THCA | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCS | |||||||
| UCS |
| Cancer | Type | Freq | Q-value |
|---|---|---|---|
| COAD | |||
| KIRP | |||
| LUAD | |||
| LUSC | |||
| MESO | |||
| PRAD | |||
| STAD | |||
| THCA |
| Cancer | P-value | Q-value |
|---|---|---|
| THYM | ||
| MESO | ||
| ACC | ||
| SKCM | ||
| KIRP | ||
| COAD | ||
| PCPG | ||
| READ | ||
| LAML | ||
| LGG | ||
| CHOL | ||
| LUAD |
| Protein ID | Domain | Pfam ID | E-value | Domain number | Total number |
|---|---|---|---|---|---|
| ENSP00000284049 | ResIII | PF04851.15 | 8.1e-07 | 1 | 1 |
| ENSP00000483667 | ResIII | PF04851.15 | 8.1e-07 | 1 | 1 |
| PID | Title | Article | Time | Author | Doi |
|---|---|---|---|---|---|
| 17620414 |
| Transcript ID | Name | Length | RefSeq ID | Protein ID | Length | RefSeq ID | UniportKB ID |
|---|---|---|---|---|---|---|---|
| ENST00000508756 | CHD1-205 | 724 | - | - | - (aa) | - | - |
| ENST00000614616 | CHD1-212 | 8095 | - | ENSP00000483667 | 1710 (aa) | - | O14646 |
| ENST00000511628 | CHD1-207 | 599 | - | - | - (aa) | - | - |
| ENST00000505657 | CHD1-203 | 736 | - | ENSP00000422225 | 113 (aa) | - | H0Y8V4 |
| ENST00000414220 | CHD1-202 | 434 | - | - | - (aa) | - | - |
| ENST00000284049 | CHD1-201 | 6457 | XM_005271866 | ENSP00000284049 | 1710 (aa) | XP_005271924 | O14646 |
| ENST00000505982 | CHD1-204 | 758 | - | - | - (aa) | - | - |
| ENST00000512392 | CHD1-208 | 1968 | - | - | - (aa) | - | - |
| ENST00000512844 | CHD1-209 | 848 | - | ENSP00000422589 | 22 (aa) | - | H0Y8Z0 |
| ENST00000514344 | CHD1-211 | 2955 | - | - | - (aa) | - | - |
| ENST00000513064 | CHD1-210 | 786 | - | - | - (aa) | - | - |
| ENST00000511067 | CHD1-206 | 817 | - | ENSP00000479403 | 272 (aa) | - | A0A087WVF4 |
| ensgID | SNP | Chromosome | Position | SNP-risk | Trait | PubmedID | 95% CI | Or or BEAT | EFO ID |
|---|---|---|---|---|---|---|---|---|---|
| ENSG00000153922 | rs11317778 | 5 | 98905525 | T | Neutrophil percentage of granulocytes | 27863252 | [0.02-0.036] unit increase | 0.02782362 | EFO_0007994 |
| ENSG00000153922 | rs11317778 | 5 | 98905525 | T | Eosinophil percentage of granulocytes | 27863252 | [0.022-0.037] unit decrease | 0.0293293 | EFO_0007996 |
| ENSG00000153922 | rs11317778 | 5 | 98905525 | T | Eosinophil percentage of white cells | 27863252 | [0.022-0.037] unit decrease | 0.02928997 | EFO_0007991 |
| ENSG00000153922 | rs11317778 | 5 | 98905525 | T | Eosinophil counts | 27863252 | [0.018-0.033] unit decrease | 0.02551863 | EFO_0004842 |
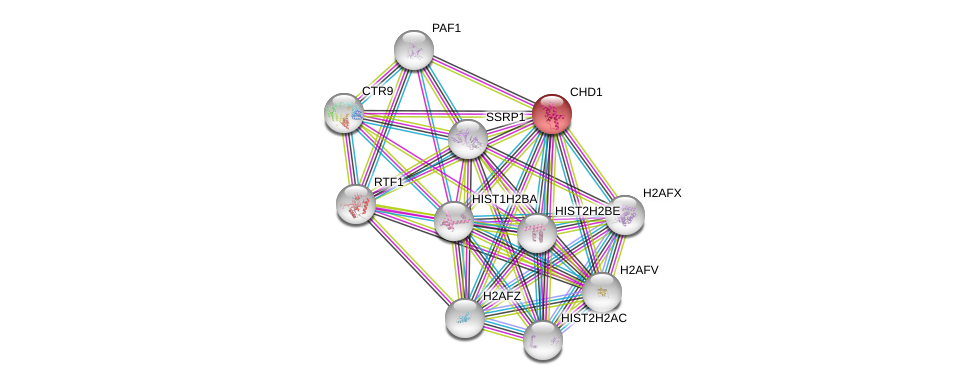
| Ensembl ID | Gene Symbol | Coverage | Identiy | Paralog | Gene Symbol | Coverage | Identiy |
|---|---|---|---|---|---|---|---|
| ENSG00000153922 | CHD1 | 95 | 57.023 | ENSG00000173575 | CHD2 | 87 | 94.898 |
| Ensembl ID | Gene Symbol | Coverage | Identiy | Ortholog | Gene Symbol | Coverage | Identiy | Species |
|---|---|---|---|---|---|---|---|---|
| ENSG00000153922 | CHD1 | 67 | 82.627 | ENSAPOG00000005764 | chd1 | 97 | 82.971 | Acanthochromis_polyacanthus |
| ENSG00000153922 | CHD1 | 95 | 83.178 | ENSAOCG00000023105 | chd1 | 94 | 77.531 | Amphiprion_ocellaris |
| ENSG00000153922 | CHD1 | 95 | 83.178 | ENSAPEG00000013544 | chd1 | 94 | 78.721 | Amphiprion_percula |
| ENSG00000153922 | CHD1 | 71 | 82.319 | ENSAMXG00000010843 | chd1 | 91 | 83.502 | Astyanax_mexicanus |
| ENSG00000153922 | CHD1 | 100 | 99.299 | ENSCATG00000032035 | CHD1 | 100 | 99.299 | Cercocebus_atys |
| ENSG00000153922 | CHD1 | 100 | 99.241 | ENSCSAG00000015339 | CHD1 | 100 | 99.241 | Chlorocebus_sabaeus |
| ENSG00000153922 | CHD1 | 100 | 97.487 | ENSCANG00000036916 | CHD1 | 100 | 97.487 | Colobus_angolensis_palliatus |
| ENSG00000153922 | CHD1 | 100 | 96.145 | ENSCGRG00001023943 | Chd1 | 100 | 96.145 | Cricetulus_griseus_chok1gshd |
| ENSG00000153922 | CHD1 | 100 | 96.145 | ENSCGRG00000008652 | Chd1 | 100 | 96.145 | Cricetulus_griseus_crigri |
| ENSG00000153922 | CHD1 | 96 | 79.817 | ENSCSEG00000009553 | chd1 | 92 | 77.435 | Cynoglossus_semilaevis |
| ENSG00000153922 | CHD1 | 94 | 92.453 | ENSEASG00005016110 | CHD1 | 99 | 97.923 | Equus_asinus_asinus |
| ENSG00000153922 | CHD1 | 96 | 80.734 | ENSELUG00000017677 | chd1 | 93 | 78.115 | Esox_lucius |
| ENSG00000153922 | CHD1 | 71 | 81.255 | ENSFHEG00000021164 | chd1 | 87 | 82.759 | Fundulus_heteroclitus |
| ENSG00000153922 | CHD1 | 91 | 84.466 | ENSGMOG00000001537 | chd1 | 96 | 67.184 | Gadus_morhua |
| ENSG00000153922 | CHD1 | 91 | 89.994 | ENSGALG00000031327 | - | 96 | 90.121 | Gallus_gallus |
| ENSG00000153922 | CHD1 | 96 | 72.477 | ENSGAFG00000019880 | chd1 | 92 | 82.844 | Gambusia_affinis |
| ENSG00000153922 | CHD1 | 99 | 96.226 | ENSGGOG00000011463 | CHD1 | 99 | 96.226 | Gorilla_gorilla |
| ENSG00000153922 | CHD1 | 91 | 84.466 | ENSHCOG00000014222 | chd1 | 91 | 79.678 | Hippocampus_comes |
| ENSG00000153922 | CHD1 | 91 | 83.495 | ENSIPUG00000001369 | chd1 | 92 | 81.380 | Ictalurus_punctatus |
| ENSG00000153922 | CHD1 | 63 | 95.154 | ENSLAFG00000023183 | CHD1 | 99 | 95.154 | Loxodonta_africana |
| ENSG00000153922 | CHD1 | 100 | 99.299 | ENSMFAG00000031226 | CHD1 | 100 | 99.299 | Macaca_fascicularis |
| ENSG00000153922 | CHD1 | 83 | 99.085 | ENSMMUG00000014090 | CHD1 | 99 | 99.085 | Macaca_mulatta |
| ENSG00000153922 | CHD1 | 100 | 99.241 | ENSMNEG00000045073 | CHD1 | 100 | 99.241 | Macaca_nemestrina |
| ENSG00000153922 | CHD1 | 99 | 95.275 | ENSMLEG00000041096 | CHD1 | 99 | 95.688 | Mandrillus_leucophaeus |
| ENSG00000153922 | CHD1 | 72 | 80.565 | ENSMZEG00005010957 | chd1 | 89 | 83.572 | Maylandia_zebra |
| ENSG00000153922 | CHD1 | 100 | 93.341 | ENSMOCG00000019121 | Chd1 | 100 | 93.224 | Microtus_ochrogaster |
| ENSG00000153922 | CHD1 | 79 | 78.267 | ENSMALG00000013317 | chd1 | 95 | 78.072 | Monopterus_albus |
| ENSG00000153922 | CHD1 | 100 | 94.454 | MGP_CAROLIEiJ_G0021099 | Chd1 | 99 | 96.852 | Mus_caroli |
| ENSG00000153922 | CHD1 | 100 | 95.738 | ENSMUSG00000023852 | Chd1 | 99 | 97.037 | Mus_musculus |
| ENSG00000153922 | CHD1 | 100 | 94.396 | MGP_PahariEiJ_G0023414 | Chd1 | 99 | 97.040 | Mus_pahari |
| ENSG00000153922 | CHD1 | 89 | 76.076 | ENSNBRG00000006860 | chd1 | 91 | 77.741 | Neolamprologus_brichardi |
| ENSG00000153922 | CHD1 | 99 | 96.952 | ENSNLEG00000014493 | CHD1 | 99 | 96.952 | Nomascus_leucogenys |
| ENSG00000153922 | CHD1 | 100 | 93.031 | ENSOANG00000004891 | CHD1 | 100 | 94.394 | Ornithorhynchus_anatinus |
| ENSG00000153922 | CHD1 | 59 | 76.794 | ENSORLG00015016725 | chd1 | 93 | 77.223 | Oryzias_latipes_hsok |
| ENSG00000153922 | CHD1 | 99 | 98.294 | ENSPPAG00000043367 | CHD1 | 99 | 98.294 | Pan_paniscus |
| ENSG00000153922 | CHD1 | 100 | 99.708 | ENSPTRG00000017103 | CHD1 | 100 | 99.708 | Pan_troglodytes |
| ENSG00000153922 | CHD1 | 100 | 99.299 | ENSPANG00000006387 | CHD1 | 100 | 99.299 | Papio_anubis |
| ENSG00000153922 | CHD1 | 94 | 83.019 | ENSPKIG00000024719 | chd1 | 92 | 79.134 | Paramormyrops_kingsleyae |
| ENSG00000153922 | CHD1 | 73 | 81.315 | ENSPMGG00000005785 | chd1 | 93 | 81.315 | Periophthalmus_magnuspinnatus |
| ENSG00000153922 | CHD1 | 100 | 95.911 | ENSPEMG00000015939 | Chd1 | 100 | 95.911 | Peromyscus_maniculatus_bairdii |
| ENSG00000153922 | CHD1 | 50 | 76.533 | ENSPLAG00000006473 | - | 85 | 77.765 | Poecilia_latipinna |
| ENSG00000153922 | CHD1 | 96 | 74.265 | ENSPREG00000004209 | chd1 | 97 | 74.759 | Poecilia_reticulata |
| ENSG00000153922 | CHD1 | 83 | 95.701 | ENSRNOG00000014434 | Chd1 | 99 | 95.490 | Rattus_norvegicus |
| ENSG00000153922 | CHD1 | 100 | 96.264 | ENSRBIG00000044325 | CHD1 | 100 | 96.439 | Rhinopithecus_bieti |
| ENSG00000153922 | CHD1 | 100 | 99.124 | ENSRROG00000001726 | CHD1 | 100 | 99.124 | Rhinopithecus_roxellana |
| ENSG00000153922 | CHD1 | 71 | 81.766 | ENSSFOG00015007161 | chd1 | 90 | 82.439 | Scleropages_formosus |
| ENSG00000153922 | CHD1 | 79 | 78.335 | ENSSDUG00000018314 | chd1 | 94 | 77.966 | Seriola_dumerili |
| ENSG00000153922 | CHD1 | 51 | 83.713 | ENSSLDG00000017244 | chd1 | 84 | 86.019 | Seriola_lalandi_dorsalis |
| ENSG00000153922 | CHD1 | 79 | 78.835 | ENSSPAG00000007024 | chd1 | 95 | 78.171 | Stegastes_partitus |
| ENSG00000153922 | CHD1 | 92 | 96.154 | ENSSSCG00000014177 | CHD1 | 100 | 98.106 | Sus_scrofa |
| ENSG00000153922 | CHD1 | 90 | 75.418 | ENSTRUG00000010282 | chd1 | 96 | 73.418 | Takifugu_rubripes |
| ENSG00000153922 | CHD1 | 72 | 77.606 | ENSTNIG00000007101 | chd1 | 95 | 78.204 | Tetraodon_nigroviridis |
| ENSG00000153922 | CHD1 | 96 | 73.394 | ENSXCOG00000017184 | chd1 | 91 | 75.244 | Xiphophorus_couchianus |
| ENSG00000153922 | CHD1 | 77 | 77.736 | ENSXMAG00000014538 | chd1 | 95 | 78.621 | Xiphophorus_maculatus |
| Go ID | Go_term | PubmedID | Evidence | Category |
|---|---|---|---|---|
| GO:0001650 | fibrillar center | - | IDA | Component |
| GO:0003677 | DNA binding | - | IEA | Function |
| GO:0004003 | ATP-dependent DNA helicase activity | 9326634. | TAS | Function |
| GO:0005515 | protein binding | 22419161.26751641. | IPI | Function |
| GO:0005524 | ATP binding | - | IEA | Function |
| GO:0005634 | nucleus | 22046413. | IDA | Component |
| GO:0005654 | nucleoplasm | - | TAS | Component |
| GO:0005737 | cytoplasm | 22046413. | IDA | Component |
| GO:0006338 | chromatin remodeling | - | IEA | Process |
| GO:0006357 | regulation of transcription by RNA polymerase II | 9326634. | TAS | Process |
| GO:0016569 | covalent chromatin modification | 28866611. | IMP | Process |
| GO:0032508 | DNA duplex unwinding | - | IEA | Process |
| GO:0035064 | methylated histone binding | 21029866. | IDA | Function |
| GO:0035064 | methylated histone binding | 28866611. | IMP | Function |
| GO:0043923 | positive regulation by host of viral transcription | 25297984. | IMP | Process |