
| Cancer | Chr | Position | Mutation Type | dbSNP | Protein-change | Allele Freq | RBD |
|---|---|---|---|---|---|---|---|
| ACC | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CHOL | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| ESCA | |||||||
| ESCA | |||||||
| ESCA | |||||||
| ESCA | |||||||
| ESCA | |||||||
| ESCA | |||||||
| ESCA | |||||||
| ESCA | |||||||
| ESCA | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| KICH | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRP | |||||||
| LGG | |||||||
| LGG | |||||||
| LGG | |||||||
| LGG | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| PAAD | |||||||
| PAAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| THCA | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCS | |||||||
| UCS |
| Cancer | Type | Freq | Q-value |
|---|---|---|---|
| DLBC | |||
| ESCA | |||
| KIRC | |||
| PAAD | |||
| READ | |||
| SKCM | |||
| TGCT | |||
| THCA |
| Cancer | P-value | Q-value |
|---|---|---|
| KIRC | ||
| SARC | ||
| UCS | ||
| SKCM | ||
| BRCA | ||
| KIRP | ||
| PAAD | ||
| CESC | ||
| LGG | ||
| UVM | ||
| OV |
| Transcript ID | Name | Length | RefSeq ID | Protein ID | Length | RefSeq ID | UniportKB ID |
|---|---|---|---|---|---|---|---|
| ENST00000368131 | IFI16-204 | 2734 | XM_017001150 | ENSP00000357113 | 729 (aa) | XP_016856639 | Q16666 |
| ENST00000368132 | IFI16-205 | 2704 | - | ENSP00000357114 | 729 (aa) | - | Q16666 |
| ENST00000493884 | IFI16-211 | 4138 | - | - | - (aa) | - | - |
| ENST00000447473 | IFI16-207 | 951 | - | ENSP00000407052 | 180 (aa) | - | X6RHM1 |
| ENST00000295809 | IFI16-201 | 2867 | XM_005245127 | ENSP00000295809 | 785 (aa) | - | Q16666 |
| ENST00000562225 | IFI16-212 | 1108 | - | - | - (aa) | - | - |
| ENST00000567661 | IFI16-214 | 546 | - | ENSP00000456094 | 74 (aa) | - | H3BR65 |
| ENST00000483916 | IFI16-210 | 4138 | - | - | - (aa) | - | - |
| ENST00000340979 | IFI16-202 | 2732 | - | ENSP00000342741 | 243 (aa) | - | A0A0A0MRB1 |
| ENST00000448393 | IFI16-208 | 2245 | XM_006711290 | ENSP00000404325 | 673 (aa) | XP_006711353 | Q16666 |
| ENST00000566111 | IFI16-213 | 856 | - | ENSP00000458084 | 83 (aa) | - | H3BVE6 |
| ENST00000359709 | IFI16-203 | 2609 | - | ENSP00000352740 | 729 (aa) | - | Q16666 |
| ENST00000426592 | IFI16-206 | 761 | - | ENSP00000406406 | 167 (aa) | - | H3BM18 |
| ENST00000474473 | IFI16-209 | 1095 | - | ENSP00000456129 | 229 (aa) | - | H3BR88 |
| Pathway ID | Pathway Name | Source |
|---|---|---|
| hsa04621 | NOD-like receptor signaling pathway | KEGG |
| ensgID | Trait | pValue | Pubmed ID |
|---|---|---|---|
| ENSG00000163565 | Platelet Function Tests | 4.2500000E-007 | - |
| ENSG00000163565 | Platelet Function Tests | 3.4000000E-006 | - |
| ENSG00000163565 | Blood Pressure | 9.2912734E-005 | - |
| ENSG00000163565 | Blood Pressure | 1.3910835E-007 | - |
| ENSG00000163565 | Leukocyte Count | 5E-47 | 23263863 |
| ENSG00000163565 | Autoimmune Diseases | 8E-7 | 21383967 |
| ENSG00000163565 | Energy Intake | 1E-6 | 23251661 |
| ENSG00000163565 | Periodontitis | 3E-8 | 26962152 |
| ENSG00000163565 | Tooth | 3E-8 | 26962152 |
| ENSG00000163565 | Neutrophils | 3E-6 | 25884002 |
| ENSG00000163565 | HIV-1 | 3E-6 | 25884002 |
| ensgID | SNP | Chromosome | Position | SNP-risk | Trait | PubmedID | 95% CI | Or or BEAT | EFO ID |
|---|---|---|---|---|---|---|---|---|---|
| ENSG00000163565 | rs1633266 | 1 | 159036187 | ? | Periodontal disease-related phenotypes | 26962152 | [0.6-1.26] unit increase | 0.93 | EFO_0000649|EFO_0007780 |
| ENSG00000163565 | rs2570916 | 1 | 159042856 | ? | Neutrophil count in HIV-infection | 25884002 | [NR] unit decrease | 0.041722316 | EFO_0004833|EFO_0000180 |
| ENSG00000163565 | rs4657616 | 1 | 159001296 | G | Hematology traits | 23263863 | [0.053-0.07] unit increase | 0.0612 | EFO_0004308 |
| ENSG00000163565 | rs861318 | 1 | 159032432 | A | Obesity-related traits | 23251661 | [NR] g/d increase | 0.04 | EFO_0003939 |
| ENSG00000163565 | rs72709516 | 1 | 159035061 | T | Blood protein levels | 29875488 | [0.73-0.97] unit increase | 0.85 | EFO_0007937 |
| ENSG00000163565 | rs12727764 | 1 | 159012687 | G | Monocyte chemoattractant protein-1 levels | 27989323 | [0.055-0.139] SD units decrease | 0.0971 | EFO_0004749 |
| ENSG00000163565 | rs1772408 | 1 | 159035859 | ? | Celiac disease or Rheumatoid arthritis | 21383967 | EFO_0000540 |
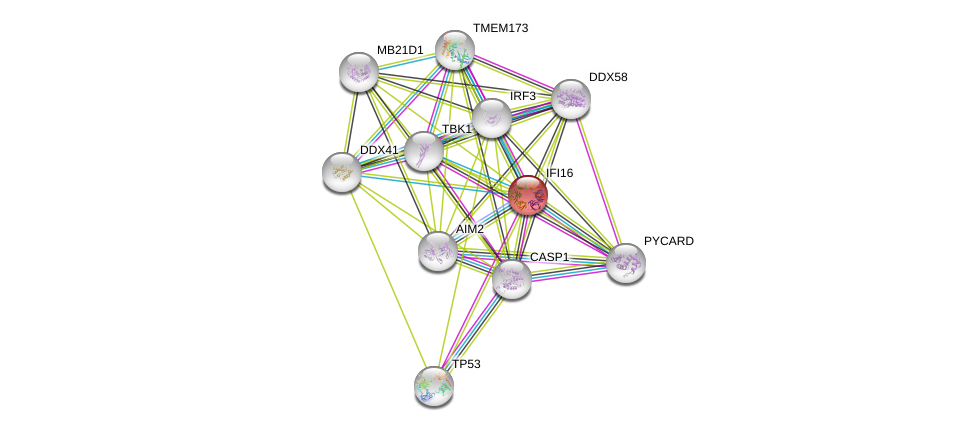
| Ensembl ID | Gene Symbol | Coverage | Identiy | Ortholog | Gene Symbol | Coverage | Identiy | Species |
|---|---|---|---|---|---|---|---|---|
| ENSG00000163565 | IFI16 | 95 | 62.025 | ENSMUSG00000090272 | Mndal | 84 | 61.628 | Mus_musculus |
| ENSG00000163565 | IFI16 | 85 | 41.546 | ENSMUSG00000037860 | Aim2 | 100 | 42.029 | Mus_musculus |
| Go ID | Go_term | PubmedID | Evidence | Category |
|---|---|---|---|---|
| GO:0000122 | negative regulation of transcription by RNA polymerase II | 12894224.24413532. | IDA | Process |
| GO:0000122 | negative regulation of transcription by RNA polymerase II | 22046441. | IMP | Process |
| GO:0000978 | RNA polymerase II proximal promoter sequence-specific DNA binding | 24413532. | IDA | Function |
| GO:0001227 | DNA-binding transcription repressor activity, RNA polymerase II-specific | 24413532. | IDA | Function |
| GO:0001819 | positive regulation of cytokine production | 22046441. | TAS | Process |
| GO:0002218 | activation of innate immune response | 21575908. | IDA | Process |
| GO:0003690 | double-stranded DNA binding | 7536752. | IDA | Function |
| GO:0003723 | RNA binding | 22658674. | HDA | Function |
| GO:0005515 | protein binding | 11146555.14654789.16494870.20890285.21397192.24413532.24531343. | IPI | Function |
| GO:0005634 | nucleus | 7536752.19158679.24413532.24531343. | IDA | Component |
| GO:0005654 | nucleoplasm | 14654789. | IDA | Component |
| GO:0005730 | nucleolus | 14654789. | IDA | Component |
| GO:0005829 | cytosol | - | IDA | Component |
| GO:0005829 | cytosol | 24531343. | IPI | Component |
| GO:0005829 | cytosol | - | TAS | Component |
| GO:0006914 | autophagy | - | IEA | Process |
| GO:0006954 | inflammatory response | - | IEA | Process |
| GO:0008134 | transcription factor binding | 12894224.22291595. | IDA | Function |
| GO:0008283 | cell proliferation | 9766636. | NAS | Process |
| GO:0010506 | regulation of autophagy | 21573174. | IEP | Process |
| GO:0016020 | membrane | 19946888. | HDA | Component |
| GO:0016607 | nuclear speck | 19158679. | IDA | Component |
| GO:0030097 | hemopoiesis | 9766636. | NAS | Process |
| GO:0030099 | myeloid cell differentiation | 7536752. | NAS | Process |
| GO:0030224 | monocyte differentiation | 9766636. | IDA | Process |
| GO:0032481 | positive regulation of type I interferon production | - | TAS | Process |
| GO:0032731 | positive regulation of interleukin-1 beta production | 21575908. | IDA | Process |
| GO:0035458 | cellular response to interferon-beta | - | IEA | Process |
| GO:0040029 | regulation of gene expression, epigenetic | 24413532. | IMP | Process |
| GO:0042149 | cellular response to glucose starvation | 21573174. | IDA | Process |
| GO:0042771 | intrinsic apoptotic signaling pathway in response to DNA damage by p53 class mediator | 14654789. | IDA | Process |
| GO:0043392 | negative regulation of DNA binding | 22291595. | IDA | Process |
| GO:0045071 | negative regulation of viral genome replication | 22291595. | IDA | Process |
| GO:0045087 | innate immune response | - | IEA | Process |
| GO:0045824 | negative regulation of innate immune response | 22046441. | IDA | Process |
| GO:0045892 | negative regulation of transcription, DNA-templated | 9642285. | IDA | Process |
| GO:0045944 | positive regulation of transcription by RNA polymerase II | 11146555. | IDA | Process |
| GO:0045944 | positive regulation of transcription by RNA polymerase II | 21575908. | IMP | Process |
| GO:0051607 | defense response to virus | 21478870. | IDA | Process |
| GO:0051607 | defense response to virus | 21575908. | IMP | Process |
| GO:0071479 | cellular response to ionizing radiation | 14654789. | IDA | Process |
| GO:0072332 | intrinsic apoptotic signaling pathway by p53 class mediator | 21573174. | IMP | Process |
| GO:0097202 | activation of cysteine-type endopeptidase activity | 21575908. | IMP | Process |
| GO:2000117 | negative regulation of cysteine-type endopeptidase activity | 22046441. | IDA | Process |