
| Cancer | Chr | Position | Mutation Type | dbSNP | Protein-change | Allele Freq | RBD |
|---|---|---|---|---|---|---|---|
| ACC | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CHOL | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| DLBC | |||||||
| DLBC | |||||||
| ESCA | |||||||
| ESCA | |||||||
| ESCA | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| LAML | |||||||
| LGG | |||||||
| LGG | |||||||
| LGG | |||||||
| LGG | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| OV | |||||||
| OV | |||||||
| PRAD | |||||||
| PRAD | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| SARC | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| THYM | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC |
| Cancer | Type | Freq | Q-value |
|---|---|---|---|
| ACC | |||
| BRCA | |||
| CHOL | |||
| COAD | |||
| ESCA | |||
| GBM | |||
| KIRC | |||
| KIRP | |||
| LGG | |||
| LIHC | |||
| MESO | |||
| OV | |||
| PAAD | |||
| READ | |||
| SARC | |||
| SKCM | |||
| STAD |
| Cancer | P-value | Q-value |
|---|---|---|
| KIRC | ||
| SARC | ||
| ACC | ||
| HNSC | ||
| PRAD | ||
| KIRP | ||
| PCPG | ||
| BLCA | ||
| CESC | ||
| LAML | ||
| LGG | ||
| CHOL |
| Transcript ID | Name | Length | RefSeq ID | Protein ID | Length | RefSeq ID | UniportKB ID |
|---|---|---|---|---|---|---|---|
| ENST00000555456 | GPHN-207 | 841 | - | ENSP00000450706 | 280 (aa) | - | H0YJ30 |
| ENST00000556240 | GPHN-212 | 404 | - | ENSP00000450442 | 60 (aa) | - | H0YIY4 |
| ENST00000557654 | GPHN-215 | 503 | - | ENSP00000451790 | 49 (aa) | - | G3V4R0 |
| ENST00000478722 | GPHN-203 | 4297 | XM_011536342 | ENSP00000417901 | 769 (aa) | XP_011534644 | Q9NQX3 |
| ENST00000459628 | GPHN-202 | 1723 | XM_017020925 | ENSP00000452220 | 380 (aa) | XP_016876414 | G3V582 |
| ENST00000315266 | GPHN-201 | 4193 | XM_005267254 | ENSP00000312771 | 736 (aa) | XP_005267311 | Q9NQX3 |
| ENST00000543237 | GPHN-204 | 2459 | XM_011536340 | ENSP00000438404 | 782 (aa) | XP_011534642 | F5H039 |
| ENST00000557678 | GPHN-216 | 592 | - | - | - (aa) | - | - |
| ENST00000555527 | GPHN-209 | 493 | - | - | - (aa) | - | - |
| ENST00000544752 | GPHN-205 | 2514 | - | - | - (aa) | - | - |
| ENST00000555668 | GPHN-210 | 550 | - | ENSP00000451935 | 49 (aa) | - | G3V4R0 |
| ENST00000556633 | GPHN-214 | 512 | - | ENSP00000451056 | 50 (aa) | - | G3V355 |
| ENST00000555503 | GPHN-208 | 655 | - | ENSP00000452009 | 201 (aa) | - | H0YJR5 |
| ENST00000556020 | GPHN-211 | 562 | - | - | - (aa) | - | - |
| ENST00000553936 | GPHN-206 | 574 | - | ENSP00000451725 | 39 (aa) | - | G3V4D2 |
| ENST00000556501 | GPHN-213 | 435 | - | - | - (aa) | - | - |
| ensgID | Trait | pValue | Pubmed ID |
|---|---|---|---|
| ENSG00000171723 | Blood Pressure | 1.82739188185899E-5 | 17903302 |
| ENSG00000171723 | Blood Pressure | 6.69140255928902E-5 | 17903302 |
| ENSG00000171723 | Echocardiography | 4.90727159327964E-5 | 17903301 |
| ENSG00000171723 | Echocardiography | 8.60119811645355E-5 | 17903301 |
| ENSG00000171723 | Body Height | 9.9290000E-005 | - |
| ENSG00000171723 | Platelet Function Tests | 4.4408920E-016 | - |
| ENSG00000171723 | Breast Neoplasms | 8.1450000E-006 | - |
| ENSG00000171723 | Breast Neoplasms | 2.6510000E-005 | - |
| ENSG00000171723 | Breast Neoplasms | 6.5970000E-005 | - |
| ENSG00000171723 | Breast Neoplasms | 3.0460000E-005 | - |
| ENSG00000171723 | Breast Neoplasms | 4.0670000E-006 | - |
| ENSG00000171723 | Gait | 2E-7 | 26219847 |
| ENSG00000171723 | Depressive Disorder | 3E-6 | 23290196 |
| ensgID | SNP | Chromosome | Position | SNP-risk | Trait | PubmedID | 95% CI | Or or BEAT | EFO ID |
|---|---|---|---|---|---|---|---|---|---|
| ENSG00000171723 | rs146647518 | 14 | 67062903 | A | Tandem gait | 26219847 | [5.12-11.24] unit increase | 8.183 | EFO_0007680 |
| ENSG00000171723 | rs8020095 | 14 | 66987141 | A | Depression (quantitative trait) | 23290196 | EFO_0003761 | ||
| ENSG00000171723 | rs17103572 | 14 | 66519254 | ? | Adolescent idiopathic scoliosis | 30019117 | EFO_0005423 |
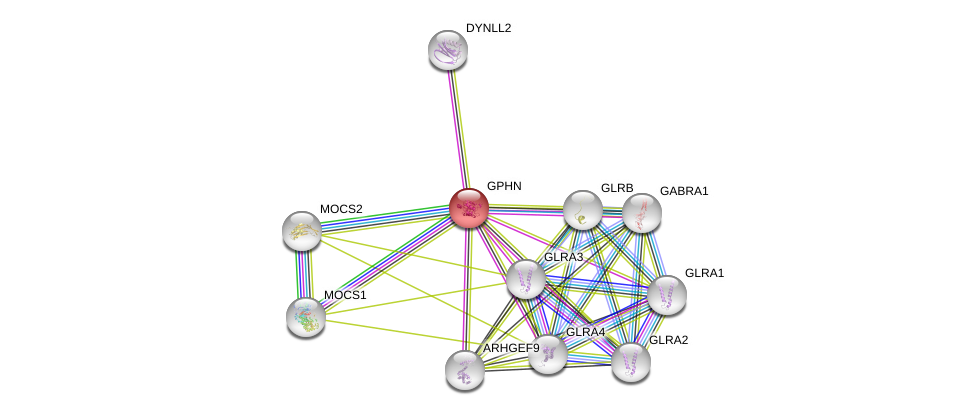
| Ensembl ID | Gene Symbol | Coverage | Identiy | Ortholog | Gene Symbol | Coverage | Identiy | Species |
|---|---|---|---|---|---|---|---|---|
| ENSG00000171723 | GPHN | 100 | 98.446 | ENSMUSG00000047454 | Gphn | 100 | 98.446 | Mus_musculus |
| Go ID | Go_term | PubmedID | Evidence | Category |
|---|---|---|---|---|
| GO:0005515 | protein binding | 21094642.26613940.27173435. | IPI | Function |
| GO:0005524 | ATP binding | - | IEA | Function |
| GO:0005737 | cytoplasm | 21873635. | IBA | Component |
| GO:0005829 | cytosol | 21873635. | IBA | Component |
| GO:0005856 | cytoskeleton | - | IEA | Component |
| GO:0005886 | plasma membrane | - | TAS | Component |
| GO:0006777 | Mo-molybdopterin cofactor biosynthetic process | 21873635. | IBA | Process |
| GO:0007529 | establishment of synaptic specificity at neuromuscular junction | 21873635. | IBA | Process |
| GO:0008940 | nitrate reductase activity | 9990024. | IMP | Function |
| GO:0010038 | response to metal ion | 9990024. | IMP | Process |
| GO:0014069 | postsynaptic density | - | ISS | Component |
| GO:0018315 | molybdenum incorporation into molybdenum-molybdopterin complex | 21873635. | IBA | Process |
| GO:0030054 | cell junction | - | IEA | Component |
| GO:0030425 | dendrite | 21873635. | IBA | Component |
| GO:0030425 | dendrite | 26613940. | IDA | Component |
| GO:0032324 | molybdopterin cofactor biosynthetic process | 21873635. | IBA | Process |
| GO:0032324 | molybdopterin cofactor biosynthetic process | 26613940. | IDA | Process |
| GO:0032324 | molybdopterin cofactor biosynthetic process | 9990024. | IMP | Process |
| GO:0032324 | molybdopterin cofactor biosynthetic process | - | TAS | Process |
| GO:0043546 | molybdopterin cofactor binding | 9990024. | IDA | Function |
| GO:0045211 | postsynaptic membrane | 21873635. | IBA | Component |
| GO:0045211 | postsynaptic membrane | 26613940. | IDA | Component |
| GO:0046872 | metal ion binding | - | IEA | Function |
| GO:0055114 | oxidation-reduction process | - | IEA | Process |
| GO:0061598 | molybdopterin adenylyltransferase activity | 21873635. | IBA | Function |
| GO:0061599 | molybdopterin molybdotransferase activity | 21873635. | IBA | Function |
| GO:0072579 | glycine receptor clustering | 21873635. | IBA | Process |
| GO:0097112 | gamma-aminobutyric acid receptor clustering | 21873635. | IBA | Process |
| GO:0097112 | gamma-aminobutyric acid receptor clustering | 26613940. | IDA | Process |
| GO:0098970 | postsynaptic neurotransmitter receptor diffusion trapping | 21873635. | IBA | Process |
| GO:0099572 | postsynaptic specialization | 21873635. | IBA | Component |
| GO:0099634 | postsynaptic specialization membrane | - | IEA | Component |