
| Cancer | Chr | Position | Mutation Type | dbSNP | Protein-change | Allele Freq | RBD |
|---|---|---|---|---|---|---|---|
| ACC | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CHOL | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| ESCA | |||||||
| ESCA | |||||||
| ESCA | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| KICH | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| LAML | |||||||
| LAML | |||||||
| LAML | |||||||
| LAML | |||||||
| LGG | |||||||
| LGG | |||||||
| LGG | |||||||
| LGG | |||||||
| LGG | |||||||
| LGG | |||||||
| LGG | |||||||
| LGG | |||||||
| LGG | |||||||
| LGG | |||||||
| LGG | |||||||
| LGG | |||||||
| LGG | |||||||
| LGG | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| MESO | |||||||
| MESO | |||||||
| MESO | |||||||
| MESO | |||||||
| MESO | |||||||
| MESO | |||||||
| MESO | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| PAAD | |||||||
| PCPG | |||||||
| PCPG | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| SARC | |||||||
| SARC | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| THCA | |||||||
| THCA | |||||||
| THYM | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCS | |||||||
| UVM |
| Cancer | Type | Freq | Q-value |
|---|---|---|---|
| CHOL | |||
| DLBC | |||
| GBM | |||
| LGG | |||
| LUAD | |||
| PAAD | |||
| SARC | |||
| SKCM | |||
| TGCT | |||
| THYM |
| Cancer | P-value | Q-value |
|---|---|---|
| THYM | ||
| KIRC | ||
| SARC | ||
| MESO | ||
| ACC | ||
| SKCM | ||
| KIRP | ||
| READ | ||
| KICH | ||
| GBM | ||
| LIHC | ||
| LGG | ||
| CHOL | ||
| LUAD | ||
| UVM |
| Transcript ID | Name | Length | RefSeq ID | Protein ID | Length | RefSeq ID | UniportKB ID |
|---|---|---|---|---|---|---|---|
| ENST00000638947 | SETD2-209 | 1984 | - | ENSP00000491413 | 591 (aa) | - | A0A1W2PPX9 |
| ENST00000409792 | SETD2-202 | 8142 | XM_011533632 | ENSP00000386759 | 2564 (aa) | XP_011531934 | Q9BYW2 |
| ENST00000492397 | SETD2-208 | 746 | - | - | - (aa) | - | - |
| ENST00000484689 | SETD2-207 | 561 | - | - | - (aa) | - | - |
| ENST00000330022 | SETD2-201 | 8172 | - | ENSP00000332415 | 1365 (aa) | - | H7BXT4 |
| ENST00000412450 | SETD2-203 | 4231 | - | ENSP00000416401 | 1340 (aa) | - | C9JG86 |
| ENST00000479832 | SETD2-206 | 794 | - | - | - (aa) | - | - |
| ENST00000431180 | SETD2-204 | 7555 | - | ENSP00000388349 | 1290 (aa) | - | H7BZ93 |
| ENST00000445387 | SETD2-205 | 7075 | - | ENSP00000411901 | 1675 (aa) | - | H7C3H4 |
| Pathway ID | Pathway Name | Source |
|---|---|---|
| hsa00310 | Lysine degradation | KEGG |
| ensgID | SNP | Chromosome | Position | SNP-risk | Trait | PubmedID | 95% CI | Or or BEAT | EFO ID |
|---|---|---|---|---|---|---|---|---|---|
| ENSG00000181555 | rs13063578 | 3 | 47046347 | A | Monocyte count | 27863252 | [0.019-0.033] unit increase | 0.02587866 | EFO_0005091 |
| ENSG00000181555 | rs151074357 | 3 | 47034978 | C | Post bronchodilator FEV1/FVC ratio | 26634245 | unit increase | 0.24 | GO_0097366|EFO_0004713 |
| ENSG00000181555 | rs2290547 | 3 | 47019693 | A | HDL cholesterol | 24097068 | [NR] unit decrease | 0.03 | EFO_0004612 |
| ENSG00000181555 | rs17784127 | 3 | 47020210 | T | Highest math class taken (MTAG) | 30038396 | [0.007-0.0136] unit increase | 0.0103 | EFO_0004875 |
| ENSG00000181555 | rs543811905 | 3 | 47029484 | CA | Granulocyte percentage of myeloid white cells | 27863252 | [0.031-0.05] unit increase | 0.04058472 | EFO_0007997 |
| ENSG00000181555 | rs13063578 | 3 | 47046347 | A | Lymphocyte counts | 27863252 | [0.048-0.062] unit increase | 0.0547414 | EFO_0004587 |
| ENSG00000181555 | rs13063578 | 3 | 47046347 | A | Lymphocyte percentage of white cells | 27863252 | [0.024-0.039] unit increase | 0.03160705 | EFO_0007993 |
| ENSG00000181555 | rs13063578 | 3 | 47046347 | A | White blood cell count (basophil) | 27863252 | [0.014-0.028] unit increase | 0.02122998 | EFO_0005090 |
| ENSG00000181555 | rs543811905 | 3 | 47029484 | CA | Platelet distribution width | 27863252 | [0.036-0.055] unit increase | 0.04532362 | EFO_0007984 |
| ENSG00000181555 | rs13063578 | 3 | 47046347 | A | White blood cell count | 27863252 | [0.026-0.04] unit increase | 0.032872 | EFO_0004308 |
| ENSG00000181555 | rs543811905 | 3 | 47029484 | CA | Monocyte percentage of white cells | 27863252 | [0.021-0.04] unit decrease | 0.0304442 | EFO_0007989 |
| ENSG00000181555 | rs111675602 | 3 | 47118447 | C | Macrophage inflammatory protein 1b levels | 27989323 | [0.2-0.31] SD units decrease | 0.2548 | EFO_0008219 |
| ENSG00000181555 | rs2290547 | 3 | 47019693 | A | HDL cholesterol levels | 28334899 | [0.021-0.039] unit decrease (EA Beta values) | 0.0297 | EFO_0004612 |
| ENSG00000181555 | rs12631730 | 3 | 47088413 | ? | Reaction time | 29844566 | [0.0049-0.0118] unit increase | 0.0083757 | EFO_0008393 |
| ENSG00000181555 | rs114151323 | 3 | 47142565 | ? | White blood cell count | 30595370 | EFO_0004308 | ||
| ENSG00000181555 | rs4082155 | 3 | 47083895 | A | Waist-to-hip ratio adjusted for BMI (additive genetic model) | 30778226 | [0.0079-0.0177] unit increase | 0.0128 | EFO_0007788 |
| ENSG00000181555 | rs4082155 | 3 | 47083895 | A | Waist-to-hip ratio adjusted for BMI (additive genetic model) | 30778226 | [0.0089-0.0227] unit increase | 0.0158 | EFO_0007788 |
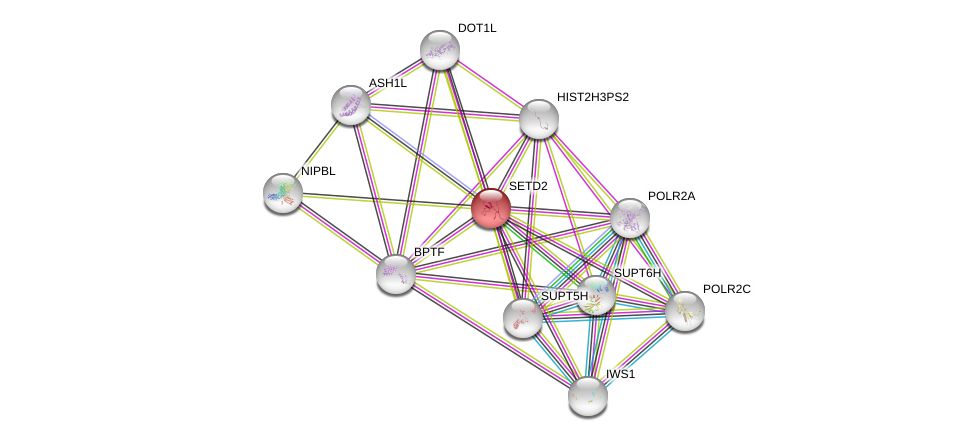
| Go ID | Go_term | PubmedID | Evidence | Category |
|---|---|---|---|---|
| GO:0001525 | angiogenesis | - | IEA | Process |
| GO:0001763 | morphogenesis of a branching structure | - | IEA | Process |
| GO:0001843 | neural tube closure | - | IEA | Process |
| GO:0005515 | protein binding | 17500595.19141475.21988832.28753426. | IPI | Function |
| GO:0005634 | nucleus | - | ISS | Component |
| GO:0005654 | nucleoplasm | - | TAS | Component |
| GO:0005694 | chromosome | - | ISS | Component |
| GO:0006298 | mismatch repair | 23622243. | IMP | Process |
| GO:0006355 | regulation of transcription, DNA-templated | - | IEA | Process |
| GO:0006368 | transcription elongation from RNA polymerase II promoter | 23325844. | IMP | Process |
| GO:0010569 | regulation of double-strand break repair via homologous recombination | 24843002. | IDA | Process |
| GO:0010793 | regulation of mRNA export from nucleus | 19141475. | IMP | Process |
| GO:0016279 | protein-lysine N-methyltransferase activity | 28753426. | IDA | Function |
| GO:0018023 | peptidyl-lysine trimethylation | 27518565. | IDA | Process |
| GO:0018024 | histone-lysine N-methyltransferase activity | 23043551. | IDA | Function |
| GO:0018024 | histone-lysine N-methyltransferase activity | 23622243. | IMP | Function |
| GO:0018024 | histone-lysine N-methyltransferase activity | - | TAS | Function |
| GO:0018026 | peptidyl-lysine monomethylation | 28753426. | IDA | Process |
| GO:0030900 | forebrain development | - | IEA | Process |
| GO:0032465 | regulation of cytokinesis | 27518565. | IDA | Process |
| GO:0032727 | positive regulation of interferon-alpha production | 28753426. | IDA | Process |
| GO:0034340 | response to type I interferon | 28753426. | IDA | Process |
| GO:0034728 | nucleosome organization | 23325844. | IMP | Process |
| GO:0035441 | cell migration involved in vasculogenesis | - | IEA | Process |
| GO:0035987 | endodermal cell differentiation | - | ISS | Process |
| GO:0043014 | alpha-tubulin binding | 27518565. | IDA | Function |
| GO:0046872 | metal ion binding | - | IEA | Function |
| GO:0046975 | histone methyltransferase activity (H3-K36 specific) | 24843002.26002201.27474439.28753426. | IDA | Function |
| GO:0048332 | mesoderm morphogenesis | - | IEA | Process |
| GO:0048701 | embryonic cranial skeleton morphogenesis | - | IEA | Process |
| GO:0048863 | stem cell differentiation | - | ISS | Process |
| GO:0048864 | stem cell development | - | IEA | Process |
| GO:0051607 | defense response to virus | 28753426. | IDA | Process |
| GO:0060039 | pericardium development | - | IEA | Process |
| GO:0060669 | embryonic placenta morphogenesis | - | IEA | Process |
| GO:0060977 | coronary vasculature morphogenesis | - | IEA | Process |
| GO:0097198 | histone H3-K36 trimethylation | 23043551.24843002.26002201.27474439.28753426. | IDA | Process |
| GO:0097198 | histone H3-K36 trimethylation | 19141475.23325844.23622243. | IMP | Process |
| GO:0097676 | histone H3-K36 dimethylation | 26002201. | IDA | Process |
| GO:1902850 | microtubule cytoskeleton organization involved in mitosis | 27518565. | IDA | Process |
| GO:1905634 | regulation of protein localization to chromatin | 24843002. | IDA | Process |