
| Cancer | Chr | Position | Mutation Type | dbSNP | Protein-change | Allele Freq | RBD |
|---|---|---|---|---|---|---|---|
| ACC | |||||||
| ACC | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| ESCA | |||||||
| ESCA | |||||||
| ESCA | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| KICH | |||||||
| KIRC | |||||||
| KIRP | |||||||
| KIRP | |||||||
| LAML | |||||||
| LGG | |||||||
| LGG | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| SARC | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| THCA | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCS |
| Cancer | Type | Freq | Q-value |
|---|---|---|---|
| ACC | |||
| COAD | |||
| ESCA | |||
| HNSC | |||
| KICH | |||
| KIRP | |||
| LUAD | |||
| LUSC | |||
| PAAD | |||
| PRAD | |||
| READ | |||
| SARC | |||
| STAD |
| Cancer | P-value | Q-value |
|---|---|---|
| THYM | ||
| KIRC | ||
| ACC | ||
| LUSC | ||
| KIRP | ||
| PAAD | ||
| BLCA | ||
| UCEC | ||
| LGG | ||
| LUAD |
| Transcript ID | Name | Length | RefSeq ID | Protein ID | Length | RefSeq ID | UniportKB ID |
|---|---|---|---|---|---|---|---|
| ENST00000612288 | CYFIP1-205 | 553 | - | ENSP00000479802 | 185 (aa) | - | A0A087WVZ5 |
| ENST00000611832 | CYFIP1-204 | 582 | - | ENSP00000478377 | 151 (aa) | - | A0A087WU52 |
| ENST00000621036 | CYFIP1-212 | 585 | - | - | - (aa) | - | - |
| ENST00000619037 | CYFIP1-209 | 575 | - | ENSP00000480596 | 77 (aa) | - | A0A087WWY5 |
| ENST00000561263 | CYFIP1-202 | 707 | - | ENSP00000453060 | 176 (aa) | - | H0YL50 |
| ENST00000622576 | CYFIP1-214 | 583 | - | ENSP00000482459 | 45 (aa) | - | A0A087WZ89 |
| ENST00000621105 | CYFIP1-213 | 514 | - | - | - (aa) | - | - |
| ENST00000617928 | CYFIP1-208 | 6793 | XM_011543873 | ENSP00000481038 | 1253 (aa) | XP_011542175 | Q7L576 |
| ENST00000613006 | CYFIP1-206 | 544 | - | ENSP00000480307 | 161 (aa) | - | A0A087WWL1 |
| ENST00000610365 | CYFIP1-203 | 4515 | XM_017022024 | ENSP00000478779 | 1253 (aa) | XP_016877513 | Q7L576 |
| ENST00000617556 | CYFIP1-207 | 5674 | - | ENSP00000480525 | 822 (aa) | - | Q7L576 |
| ENST00000619348 | CYFIP1-211 | 3471 | - | - | - (aa) | - | - |
| ENST00000557890 | CYFIP1-201 | 408 | - | - | - (aa) | - | - |
| ENST00000619290 | CYFIP1-210 | 929 | - | - | - (aa) | - | - |
| ensgID | Trait | pValue | Pubmed ID |
|---|---|---|---|
| ENSG00000273749 | E-Selectin | 4.8903000E-005 | 19729612 |
| ENSG00000273749 | Muscle, Skeletal | 8.2200000E-005 | - |
| ENSG00000273749 | Muscle, Skeletal | 1.8540000E-005 | - |
| ENSG00000273749 | Rotator Cuff | 9.9240000E-005 | - |
| ENSG00000273749 | Muscle, Skeletal | 9.1970000E-005 | - |
| ENSG00000273749 | Muscle, Skeletal | 4.6700000E-005 | - |
| ENSG00000273749 | Waist Circumference | 6.8980000E-005 | - |
| ensgID | SNP | Chromosome | Position | SNP-risk | Trait | PubmedID | 95% CI | Or or BEAT | EFO ID |
|---|---|---|---|---|---|---|---|---|---|
| ENSG00000273749 | rs7170637 | 15 | 22903836 | ? | Heel bone mineral density | 30048462 | [0.012-0.023] unit increase | 0.0172789 | EFO_0009270 |
| ENSG00000273749 | rs7170637 | 15 | 22903836 | G | Heel bone mineral density | 30598549 | [0.013-0.023] unit increase | 0.0180226 | EFO_0009270 |
| ENSG00000273749 | rs11636046 | 15 | 22949326 | ? | Red cell distribution width | 30595370 | EFO_0005192 | ||
| ENSG00000273749 | rs7170637 | 15 | 22903836 | ? | Heel bone mineral density | 30595370 | EFO_0009270 |
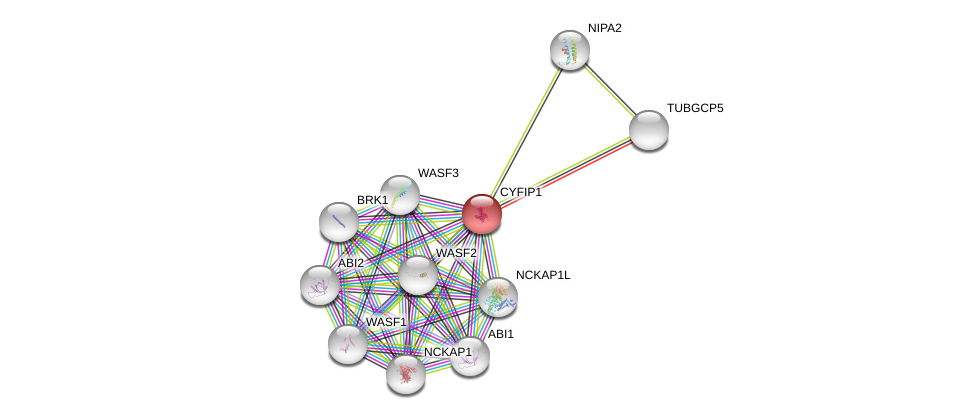
| Ensembl ID | Gene Symbol | Coverage | Identiy | Ortholog | Gene Symbol | Coverage | Identiy | Species |
|---|---|---|---|---|---|---|---|---|
| ENSG00000273749 | CYFIP1 | 100 | 92.053 | ENSG00000055163 | CYFIP2 | 100 | 93.694 | Homo_sapiens |
| Go ID | Go_term | PubmedID | Evidence | Category |
|---|---|---|---|---|
| GO:0000340 | RNA 7-methylguanosine cap binding | 21873635. | IBA | Function |
| GO:0000902 | cell morphogenesis | 21873635. | IBA | Process |
| GO:0001726 | ruffle | - | ISS | Component |
| GO:0005515 | protein binding | 11438699.15294869.15296760.16260607.18805096.19363480.21107423.24439377. | IPI | Function |
| GO:0005576 | extracellular region | - | TAS | Component |
| GO:0005829 | cytosol | - | TAS | Component |
| GO:0005845 | mRNA cap binding complex | - | ISS | Component |
| GO:0005925 | focal adhesion | 21423176. | HDA | Component |
| GO:0006417 | regulation of translation | 21873635. | IBA | Process |
| GO:0007411 | axon guidance | 21873635. | IBA | Process |
| GO:0008360 | regulation of cell shape | - | IEA | Process |
| GO:0016601 | Rac protein signal transduction | 21107423. | IMP | Process |
| GO:0030027 | lamellipodium | - | ISS | Component |
| GO:0030031 | cell projection assembly | 21873635. | IBA | Process |
| GO:0030032 | lamellipodium assembly | 21873635. | IBA | Process |
| GO:0030032 | lamellipodium assembly | - | ISS | Process |
| GO:0031209 | SCAR complex | 21873635. | IBA | Component |
| GO:0031209 | SCAR complex | 21107423. | IMP | Component |
| GO:0031529 | ruffle organization | - | ISS | Process |
| GO:0032433 | filopodium tip | - | IEA | Component |
| GO:0032869 | cellular response to insulin stimulus | - | IEA | Process |
| GO:0034774 | secretory granule lumen | - | TAS | Component |
| GO:0035580 | specific granule lumen | - | TAS | Component |
| GO:0038096 | Fc-gamma receptor signaling pathway involved in phagocytosis | - | TAS | Process |
| GO:0043005 | neuron projection | 21873635. | IBA | Component |
| GO:0043005 | neuron projection | - | ISS | Component |
| GO:0043025 | neuronal cell body | - | IEA | Component |
| GO:0043195 | terminal bouton | - | IEA | Component |
| GO:0043197 | dendritic spine | - | IEA | Component |
| GO:0043312 | neutrophil degranulation | - | TAS | Process |
| GO:0044294 | dendritic growth cone | - | IEA | Component |
| GO:0044295 | axonal growth cone | - | IEA | Component |
| GO:0045182 | translation regulator activity | - | IEA | Function |
| GO:0045202 | synapse | 21873635. | IBA | Component |
| GO:0045773 | positive regulation of axon extension | - | IEA | Process |
| GO:0048010 | vascular endothelial growth factor receptor signaling pathway | - | TAS | Process |
| GO:0048365 | Rac GTPase binding | 21107423. | IMP | Function |
| GO:0048365 | Rac GTPase binding | - | ISS | Function |
| GO:0048471 | perinuclear region of cytoplasm | - | ISS | Component |
| GO:0048675 | axon extension | 16260607. | IMP | Process |
| GO:0050890 | cognition | 17519220. | IMP | Process |
| GO:0051015 | actin filament binding | - | ISS | Function |
| GO:0051388 | positive regulation of neurotrophin TRK receptor signaling pathway | - | IEA | Process |
| GO:0051602 | response to electrical stimulus | - | IEA | Process |
| GO:0060076 | excitatory synapse | - | IEA | Component |
| GO:0070062 | extracellular exosome | 20458337.23533145. | HDA | Component |
| GO:0090724 | central region of growth cone | - | IEA | Component |
| GO:0090725 | peripheral region of growth cone | - | IEA | Component |
| GO:0097484 | dendrite extension | - | IEA | Process |
| GO:0099563 | modification of synaptic structure | 21873635. | IBA | Process |
| GO:0099578 | regulation of translation at postsynapse, modulating synaptic transmission | - | IEA | Process |
| GO:1900006 | positive regulation of dendrite development | - | IEA | Process |
| GO:1900029 | positive regulation of ruffle assembly | - | IEA | Process |
| GO:1903422 | negative regulation of synaptic vesicle recycling | - | IEA | Process |
| GO:1904724 | tertiary granule lumen | - | TAS | Component |
| GO:1905274 | regulation of modification of postsynaptic actin cytoskeleton | - | IEA | Process |
| GO:2000601 | positive regulation of Arp2/3 complex-mediated actin nucleation | 21107423. | IMP | Process |