
| Transcript ID | Name | Length | RefSeq ID | Protein ID | Length | RefSeq ID | UniportKB ID |
|---|---|---|---|---|---|---|---|
| ENST00000394419 | ACTN1-203 | 3464 | XM_017021720 | ENSP00000377941 | 914 (aa) | XP_016877209 | P12814 |
| ENST00000555075 | ACTN1-214 | 828 | - | ENSP00000452254 | 251 (aa) | - | H0YJW3 |
| ENST00000538545 | ACTN1-205 | 2813 | - | ENSP00000439828 | 930 (aa) | - | P12814 |
| ENST00000553779 | ACTN1-210 | 530 | - | ENSP00000450618 | 85 (aa) | - | G3V2E8 |
| ENST00000544964 | ACTN1-206 | 891 | - | ENSP00000444422 | 297 (aa) | - | H7C5W8 |
| ENST00000555616 | ACTN1-215 | 919 | - | ENSP00000450903 | 238 (aa) | - | G3V2W4 |
| ENST00000556083 | ACTN1-216 | 4674 | - | - | - (aa) | - | - |
| ENST00000556343 | ACTN1-218 | 274 | - | - | - (aa) | - | - |
| ENST00000556203 | ACTN1-217 | 564 | - | - | - (aa) | - | - |
| ENST00000193403 | ACTN1-201 | 3779 | XM_011537265 | ENSP00000193403 | 892 (aa) | XP_011535567 | P12814 |
| ENST00000554508 | ACTN1-213 | 585 | - | - | - (aa) | - | - |
| ENST00000553290 | ACTN1-207 | 619 | - | ENSP00000450625 | 207 (aa) | - | H0YJ11 |
| ENST00000556571 | ACTN1-221 | 568 | - | ENSP00000452423 | 126 (aa) | - | G3V5M4 |
| ENST00000438964 | ACTN1-204 | 3043 | XM_017021727 | ENSP00000414272 | 887 (aa) | XP_016877216 | P12814 |
| ENST00000553882 | ACTN1-211 | 2636 | - | - | - (aa) | - | - |
| ENST00000376839 | ACTN1-202 | 2824 | XM_011537266 | ENSP00000366035 | 822 (aa) | XP_011535568 | H9KV75 |
| ENST00000553659 | ACTN1-209 | 434 | - | ENSP00000451086 | 72 (aa) | - | G3V380 |
| ENST00000553370 | ACTN1-208 | 605 | - | ENSP00000450925 | 144 (aa) | - | G3V2X9 |
| ENST00000556432 | ACTN1-219 | 558 | - | - | - (aa) | - | - |
| ENST00000556433 | ACTN1-220 | 1259 | - | ENSP00000450764 | 260 (aa) | - | G3V2N5 |
| ENST00000554158 | ACTN1-212 | 571 | - | - | - (aa) | - | - |
| ensgID | Trait | pValue | Pubmed ID |
|---|---|---|---|
| ENSG00000072110 | Coronary Artery Disease | 8.8920000E-005 | - |
| ENSG00000072110 | Coronary Artery Disease | 8.0590000E-005 | - |
| ENSG00000072110 | Coronary Artery Disease | 7.9910000E-005 | - |
| ENSG00000072110 | Coronary Artery Disease | 5.2020000E-005 | - |
| ENSG00000072110 | Coronary Disease | 2.1020000E-005 | - |
| ENSG00000072110 | Platelet Function Tests | 1.9866100E-005 | - |
| ENSG00000072110 | Platelet Function Tests | 1.0886700E-005 | - |
| ENSG00000072110 | Platelet Function Tests | 1.3319700E-005 | - |
| ENSG00000072110 | Platelet Count | 1E-28 | 26805783 |
| ENSG00000072110 | Periodontitis | 4E-6 | 24024966 |
| ENSG00000072110 | Cognition Disorders | 2E-6 | 26252872 |
| ENSG00000072110 | Cognitive Dysfunction | 2E-6 | 26252872 |
| ENSG00000072110 | Cognition Disorders | 9E-7 | 26252872 |
| ENSG00000072110 | Cognitive Dysfunction | 9E-7 | 26252872 |
| ENSG00000072110 | Smoking | 7E-6 | 19247474 |
| ensgID | SNP | Chromosome | Position | SNP-risk | Trait | PubmedID | 95% CI | Or or BEAT | EFO ID |
|---|---|---|---|---|---|---|---|---|---|
| ENSG00000072110 | rs2073307 | 14 | 68875533 | T | Mean platelet volume | 27863252 | [0.033-0.047] unit decrease | 0.03994193 | EFO_0004584 |
| ENSG00000072110 | rs117672662 | 14 | 68958750 | C | Mean platelet volume | 27863252 | [0.36-0.52] unit increase | 0.4413049 | EFO_0004584 |
| ENSG00000072110 | rs12050161 | 14 | 68893914 | T | Periodontitis (Mean PAL) | 24024966 | [0.040-0.100] unit increase | 0.07 | EFO_0000649 |
| ENSG00000072110 | rs76745467 | 14 | 68959672 | C | Cognitive decline rate in late mild cognitive impairment | 26252872 | [0.16-0.37] unit increase | 0.2634 | HP_0100543|EFO_0007710 |
| ENSG00000072110 | rs76745467 | 14 | 68959672 | C | Cognitive decline rate in late mild cognitive impairment | 26252872 | [0.16-0.38] unit increase | 0.2707 | HP_0100543|EFO_0007710 |
| ENSG00000072110 | rs117672662 | 14 | 68958750 | T | Platelet count | 26805783 | [0.50-0.71] unit increase | 0.604 | EFO_0004309 |
| ENSG00000072110 | rs2268983 | 14 | 68941980 | ? | Smoking behavior | 19247474 | [NR] | 1.52 | EFO_0004318 |
| ENSG00000072110 | rs17835769 | 14 | 68886047 | T | Mosquito bite size | 28199695 | [0.042-0.104] unit increase | 0.0727107 | EFO_0008378 |
| ENSG00000072110 | rs4902681 | 14 | 68959900 | T | Educational attainment (MTAG) | 30038396 | [0.0055-0.0113] unit decrease | 0.0084 | EFO_0004784 |
| ENSG00000072110 | rs3784138 | 14 | 68929672 | ? | Platelet count | 29403010 | [0.022-0.04] unit decrease novel | 0.03059 | EFO_0004309 |
| ENSG00000072110 | rs117672662 | 14 | 68958750 | C | Platelet distribution width | 27863252 | [0.25-0.41] unit increase | 0.3333976 | EFO_0007984 |
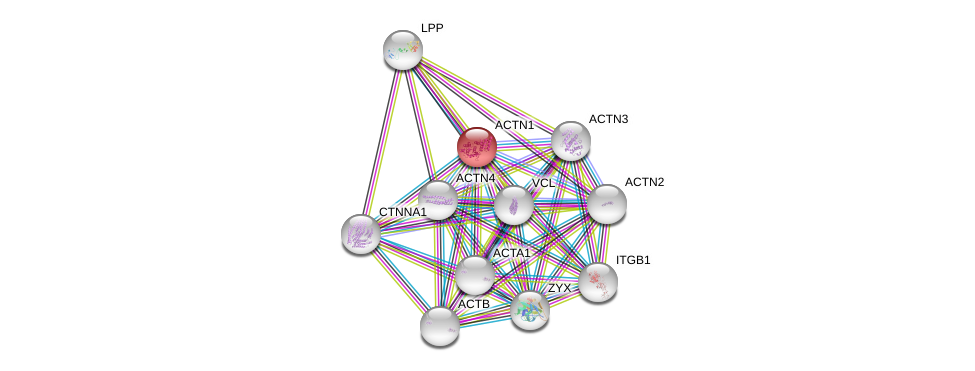
| Ensembl ID | Gene Symbol | Coverage | Identiy | Ortholog | Gene Symbol | Coverage | Identiy | Species |
|---|---|---|---|---|---|---|---|---|
| ENSG00000072110 | ACTN1 | 100 | 100.000 | ENSMUSG00000015143 | Actn1 | 100 | 99.215 | Mus_musculus |
| Go ID | Go_term | PubmedID | Evidence | Category |
|---|---|---|---|---|
| GO:0001725 | stress fiber | 11223950. | IDA | Component |
| GO:0001726 | ruffle | 11223950. | IDA | Component |
| GO:0002576 | platelet degranulation | - | TAS | Process |
| GO:0003725 | double-stranded RNA binding | 21266579. | IDA | Function |
| GO:0005178 | integrin binding | 7983147. | IDA | Function |
| GO:0005178 | integrin binding | 11223950. | IPI | Function |
| GO:0005509 | calcium ion binding | - | IEA | Function |
| GO:0005515 | protein binding | 10793131.11101506.12446711.14623081.16189514.16291744.16464232.20120020.25416956.25910212.26496610.26871637.30021884. | IPI | Function |
| GO:0005576 | extracellular region | - | TAS | Component |
| GO:0005615 | extracellular space | 16502470. | HDA | Component |
| GO:0005737 | cytoplasm | 16464232.23434115. | IDA | Component |
| GO:0005829 | cytosol | - | TAS | Component |
| GO:0005884 | actin filament | 23434115.24069336. | IDA | Component |
| GO:0005886 | plasma membrane | - | IEA | Component |
| GO:0005903 | brush border | - | IEA | Component |
| GO:0005911 | cell-cell junction | 11223950. | IDA | Component |
| GO:0005916 | fascia adherens | - | IEA | Component |
| GO:0005925 | focal adhesion | 21423176. | HDA | Component |
| GO:0005925 | focal adhesion | 11223950. | IDA | Component |
| GO:0005925 | focal adhesion | 16807302. | IMP | Component |
| GO:0007015 | actin filament organization | 23434115. | IMP | Process |
| GO:0017166 | vinculin binding | 11223950. | IDA | Function |
| GO:0017166 | vinculin binding | 15988023. | IPI | Function |
| GO:0030018 | Z disc | - | IEA | Component |
| GO:0030168 | platelet activation | 24069336. | IMP | Process |
| GO:0030220 | platelet formation | 23434115. | IMP | Process |
| GO:0030374 | nuclear receptor transcription coactivator activity | 22351778. | IDA | Function |
| GO:0031093 | platelet alpha granule lumen | - | TAS | Component |
| GO:0031143 | pseudopodium | 1629252. | TAS | Component |
| GO:0036344 | platelet morphogenesis | 23434115.24069336. | IMP | Process |
| GO:0042803 | protein homodimerization activity | 11223950. | IDA | Function |
| GO:0042981 | regulation of apoptotic process | 16807302. | NAS | Process |
| GO:0042995 | cell projection | 16464232. | IDA | Component |
| GO:0044325 | ion channel binding | 17944866.19943616. | IPI | Function |
| GO:0048041 | focal adhesion assembly | 16807302. | IMP | Process |
| GO:0051015 | actin filament binding | 11223950. | IDA | Function |
| GO:0051017 | actin filament bundle assembly | - | IEA | Process |
| GO:0051271 | negative regulation of cellular component movement | 7983147. | IMP | Process |
| GO:0051639 | actin filament network formation | 24069336. | IMP | Process |
| GO:0051764 | actin crosslink formation | - | IEA | Process |
| GO:0070062 | extracellular exosome | 19056867.19199708.21362503.23533145. | HDA | Component |
| GO:0070527 | platelet aggregation | 23434115. | IMP | Process |
| GO:0098978 | glutamatergic synapse | 29429936. | EXP | Component |
| GO:0098978 | glutamatergic synapse | 29429936. | IDA | Component |
| GO:0098978 | glutamatergic synapse | 29429936. | IMP | Component |
| GO:0099173 | postsynapse organization | - | IEA | Process |
| GO:0099186 | structural constituent of postsynapse | 29429936. | EXP | Function |
| GO:0099186 | structural constituent of postsynapse | 29429936. | IDA | Function |
| GO:0099186 | structural constituent of postsynapse | 29429936. | IMP | Function |
| GO:1903508 | positive regulation of nucleic acid-templated transcription | - | IEA | Process |
| Cancer | Chr | Position | Mutation Type | dbSNP | Protein-change | Allele Freq | RBD |
|---|---|---|---|---|---|---|---|
| ACC | |||||||
| ACC | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRP | |||||||
| LGG | |||||||
| LGG | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| PAAD | |||||||
| PAAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| SARC | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC |
| Cancer | Type | Freq | Q-value |
|---|---|---|---|
| ACC | |||
| BRCA | |||
| CHOL | |||
| COAD | |||
| GBM | |||
| KIRC | |||
| KIRP | |||
| LGG | |||
| MESO | |||
| OV | |||
| PAAD | |||
| READ | |||
| SARC |
| Cancer | P-value | Q-value |
|---|---|---|
| KIRC | ||
| STAD | ||
| MESO | ||
| ACC | ||
| HNSC | ||
| SKCM | ||
| LUSC | ||
| ESCA | ||
| KIRP | ||
| BLCA | ||
| CESC | ||
| LIHC | ||
| LUAD | ||
| UVM |