
| Protein ID | Domain | Pfam ID | E-value | Domain number | Total number |
|---|---|---|---|---|---|
| ENSP00000359361 | KH_1 | PF00013.29 | 7.9e-12 | 1 | 1 |
| ENSP00000359361 | DEAD | PF00270.29 | 2.7e-43 | 1 | 1 |
| PID | Title | Method | Time | Author | Doi |
|---|---|---|---|---|---|
| 30528433 |
| PID | Title | Article | Time | Author | Doi |
|---|---|---|---|---|---|
| 28468824 |
| Transcript ID | Name | Length | RefSeq ID | Protein ID | Length | RefSeq ID | UniportKB ID |
|---|---|---|---|---|---|---|---|
| ENST00000464221 | DDX43-202 | 568 | - | - | - (aa) | - | - |
| ENST00000370336 | DDX43-201 | 2513 | - | ENSP00000359361 | 648 (aa) | - | Q9NXZ2 |
| ENST00000479773 | DDX43-203 | 652 | - | - | - (aa) | - | - |
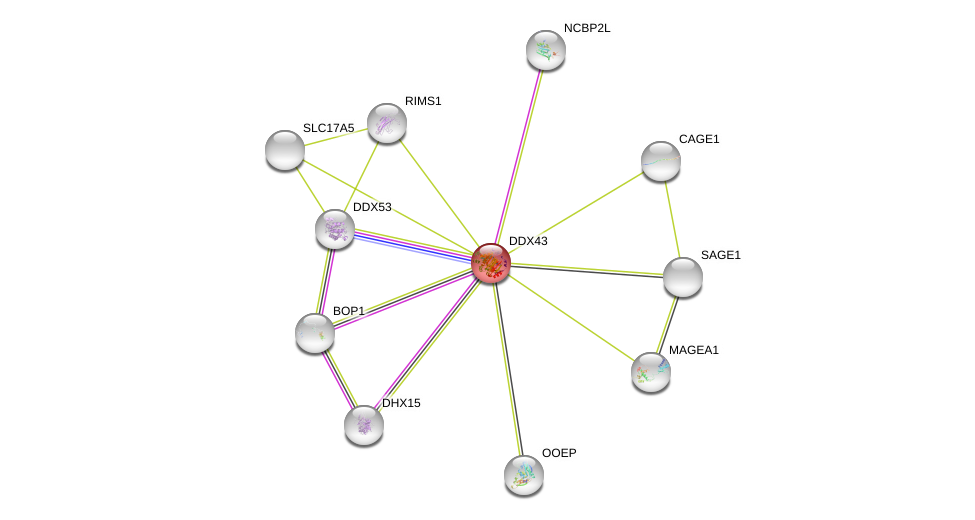
| Ensembl ID | Gene Symbol | Coverage | Identiy | Paralog | Gene Symbol | Coverage | Identiy |
|---|---|---|---|---|---|---|---|
| ENSG00000080007 | DDX43 | 55 | 33.241 | ENSG00000110367 | DDX6 | 73 | 33.241 |
| ENSG00000080007 | DDX43 | 60 | 32.857 | ENSG00000123064 | DDX54 | 90 | 39.175 |
| ENSG00000080007 | DDX43 | 61 | 37.164 | ENSG00000183258 | DDX41 | 80 | 34.752 |
| ENSG00000080007 | DDX43 | 57 | 34.133 | ENSG00000156976 | EIF4A2 | 90 | 34.133 |
| ENSG00000080007 | DDX43 | 68 | 33.260 | ENSG00000278053 | DDX52 | 77 | 32.427 |
| ENSG00000080007 | DDX43 | 56 | 34.877 | ENSG00000141543 | EIF4A3 | 87 | 34.877 |
| ENSG00000080007 | DDX43 | 56 | 30.534 | ENSG00000182810 | DDX28 | 73 | 30.534 |
| ENSG00000080007 | DDX43 | 53 | 31.500 | ENSG00000125485 | DDX31 | 53 | 31.500 |
| ENSG00000080007 | DDX43 | 51 | 35.608 | ENSG00000088205 | DDX18 | 82 | 33.846 |
| ENSG00000080007 | DDX43 | 61 | 33.741 | ENSG00000105671 | DDX49 | 82 | 33.741 |
| ENSG00000080007 | DDX43 | 68 | 42.637 | ENSG00000100201 | DDX17 | 63 | 57.143 |
| ENSG00000080007 | DDX43 | 63 | 35.194 | ENSG00000213782 | DDX47 | 86 | 35.194 |
| ENSG00000080007 | DDX43 | 56 | 36.512 | ENSG00000124228 | DDX27 | 84 | 32.618 |
| ENSG00000080007 | DDX43 | 53 | 31.856 | ENSG00000111364 | DDX55 | 87 | 35.119 |
| ENSG00000080007 | DDX43 | 59 | 40.506 | ENSG00000152670 | DDX4 | 68 | 40.506 |
| ENSG00000080007 | DDX43 | 68 | 43.430 | ENSG00000108654 | DDX5 | 100 | 56.522 |
| ENSG00000080007 | DDX43 | 96 | 61.821 | ENSG00000184735 | DDX53 | 99 | 61.821 |
| ENSG00000080007 | DDX43 | 52 | 30.259 | ENSG00000109832 | DDX25 | 72 | 45.833 |
| ENSG00000080007 | DDX43 | 60 | 35.427 | ENSG00000118197 | DDX59 | 98 | 32.883 |
| ENSG00000080007 | DDX43 | 57 | 40.674 | ENSG00000067048 | DDX3Y | 58 | 40.674 |
| ENSG00000080007 | DDX43 | 60 | 39.312 | ENSG00000215301 | DDX3X | 85 | 39.312 |
| ENSG00000080007 | DDX43 | 54 | 36.240 | ENSG00000165732 | DDX21 | 50 | 36.240 |
| ENSG00000080007 | DDX43 | 59 | 34.635 | ENSG00000161960 | EIF4A1 | 96 | 36.196 |
| ENSG00000080007 | DDX43 | 62 | 41.419 | ENSG00000174243 | DDX23 | 52 | 41.419 |
| ENSG00000080007 | DDX43 | 68 | 37.389 | ENSG00000198231 | DDX42 | 62 | 37.389 |
| Ensembl ID | Gene Symbol | Coverage | Identiy | Ortholog | Gene Symbol | Coverage | Identiy | Species |
|---|---|---|---|---|---|---|---|---|
| ENSG00000080007 | DDX43 | 100 | 87.654 | ENSANAG00000028687 | DDX43 | 100 | 87.654 | Aotus_nancymaae |
| ENSG00000080007 | DDX43 | 100 | 87.809 | ENSCJAG00000009966 | DDX43 | 100 | 87.809 | Callithrix_jacchus |
| ENSG00000080007 | DDX43 | 100 | 79.353 | ENSTSYG00000003652 | DDX43 | 100 | 79.353 | Carlito_syrichta |
| ENSG00000080007 | DDX43 | 100 | 77.932 | ENSCCAG00000037349 | DDX43 | 100 | 77.932 | Cebus_capucinus |
| ENSG00000080007 | DDX43 | 100 | 96.759 | ENSCATG00000031875 | DDX43 | 100 | 96.759 | Cercocebus_atys |
| ENSG00000080007 | DDX43 | 100 | 96.308 | ENSCSAG00000013999 | DDX43 | 100 | 96.308 | Chlorocebus_sabaeus |
| ENSG00000080007 | DDX43 | 100 | 96.759 | ENSCANG00000029269 | DDX43 | 100 | 96.759 | Colobus_angolensis_palliatus |
| ENSG00000080007 | DDX43 | 100 | 98.765 | ENSGGOG00000016092 | DDX43 | 100 | 98.765 | Gorilla_gorilla |
| ENSG00000080007 | DDX43 | 100 | 96.759 | ENSMFAG00000043087 | DDX43 | 100 | 96.759 | Macaca_fascicularis |
| ENSG00000080007 | DDX43 | 100 | 96.759 | ENSMMUG00000019599 | DDX43 | 100 | 96.759 | Macaca_mulatta |
| ENSG00000080007 | DDX43 | 100 | 92.593 | ENSMNEG00000038522 | DDX43 | 100 | 92.593 | Macaca_nemestrina |
| ENSG00000080007 | DDX43 | 100 | 96.142 | ENSMLEG00000041961 | DDX43 | 100 | 96.142 | Mandrillus_leucophaeus |
| ENSG00000080007 | DDX43 | 82 | 81.132 | ENSMICG00000017145 | DDX43 | 100 | 81.132 | Microcebus_murinus |
| ENSG00000080007 | DDX43 | 100 | 96.148 | ENSNLEG00000001209 | DDX43 | 100 | 96.148 | Nomascus_leucogenys |
| ENSG00000080007 | DDX43 | 100 | 78.923 | ENSOGAG00000013660 | DDX43 | 100 | 78.923 | Otolemur_garnettii |
| ENSG00000080007 | DDX43 | 100 | 99.074 | ENSPPAG00000034199 | DDX43 | 100 | 99.074 | Pan_paniscus |
| ENSG00000080007 | DDX43 | 100 | 72.111 | ENSPPRG00000014528 | DDX43 | 99 | 72.111 | Panthera_pardus |
| ENSG00000080007 | DDX43 | 100 | 99.074 | ENSPTRG00000018344 | DDX43 | 100 | 99.074 | Pan_troglodytes |
| ENSG00000080007 | DDX43 | 100 | 96.605 | ENSPANG00000005686 | DDX43 | 100 | 96.605 | Papio_anubis |
| ENSG00000080007 | DDX43 | 100 | 97.222 | ENSPPYG00000016774 | DDX43 | 100 | 97.222 | Pongo_abelii |
| ENSG00000080007 | DDX43 | 82 | 70.998 | ENSPCOG00000020123 | DDX43 | 100 | 71.321 | Propithecus_coquereli |
| ENSG00000080007 | DDX43 | 82 | 97.164 | ENSRBIG00000043253 | DDX43 | 100 | 97.164 | Rhinopithecus_bieti |
| ENSG00000080007 | DDX43 | 100 | 95.988 | ENSRROG00000031424 | DDX43 | 100 | 95.988 | Rhinopithecus_roxellana |
| ENSG00000080007 | DDX43 | 100 | 77.623 | ENSSBOG00000024961 | DDX43 | 100 | 77.623 | Saimiri_boliviensis_boliviensis |
| Go ID | Go_term | PubmedID | Evidence | Category |
|---|---|---|---|---|
| GO:0003723 | RNA binding | - | IEA | Function |
| GO:0004004 | ATP-dependent RNA helicase activity | 10919659. | TAS | Function |
| GO:0005524 | ATP binding | - | IEA | Function |
| Cancer | Chr | Position | Mutation Type | dbSNP | Protein-change | Allele Freq | RBD |
|---|---|---|---|---|---|---|---|
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| ESCA | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| KIRP | |||||||
| KIRP | |||||||
| LAML | |||||||
| LGG | |||||||
| LGG | |||||||
| LGG | |||||||
| LGG | |||||||
| LGG | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| PAAD | |||||||
| PAAD | |||||||
| PAAD | |||||||
| PRAD | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| THCA | |||||||
| THYM | |||||||
| THYM | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCS |
| Cancer | Type | Freq | Q-value |
|---|---|---|---|
| BLCA | |||
| CHOL | |||
| ESCA | |||
| LUAD | |||
| MESO | |||
| PAAD | |||
| SARC | |||
| SKCM | |||
| STAD |
| Cancer | P-value | Q-value |
|---|---|---|
| ACC | ||
| HNSC | ||
| SKCM | ||
| BRCA | ||
| DLBC |