
| Transcript ID | Name | Length | RefSeq ID | Protein ID | Length | RefSeq ID | UniportKB ID |
|---|---|---|---|---|---|---|---|
| ENST00000428458 | PYGB-202 | 734 | - | ENSP00000395451 | 245 (aa) | - | H0Y4Z6 |
| ENST00000216962 | PYGB-201 | 4134 | - | ENSP00000216962 | 843 (aa) | - | P11216 |
| ENST00000471359 | PYGB-203 | 726 | - | - | - (aa) | - | - |
| ensgID | SNP | Chromosome | Position | SNP-risk | Trait | PubmedID | 95% CI | Or or BEAT | EFO ID |
|---|---|---|---|---|---|---|---|---|---|
| ENSG00000100994 | rs2281558 | 20 | 25259553 | T | Calcium levels | 24068962 | [0.0091-0.0209] unit increase | 0.015 | EFO_0004838 |
| ENSG00000100994 | rs139672965 | 20 | 25286264 | A | Asymmetrical dimethylarginine levels | 24159190 | [0.37-0.94] unit increase | 0.654 | EFO_0006522|EFO_0005418 |
| ENSG00000100994 | rs2281558 | 20 | 25259553 | ? | Peripheral arterial disease (traffic-related air pollution interaction) | 27082954 | [3.88-5.4] | 4.64 | EFO_0004265|EFO_0007908 |
| ENSG00000100994 | rs73598373 | 20 | 25294985 | C | Telomere length | 30185882 | [0.133-0.311] unit decrease | 0.222 | EFO_0004505 |
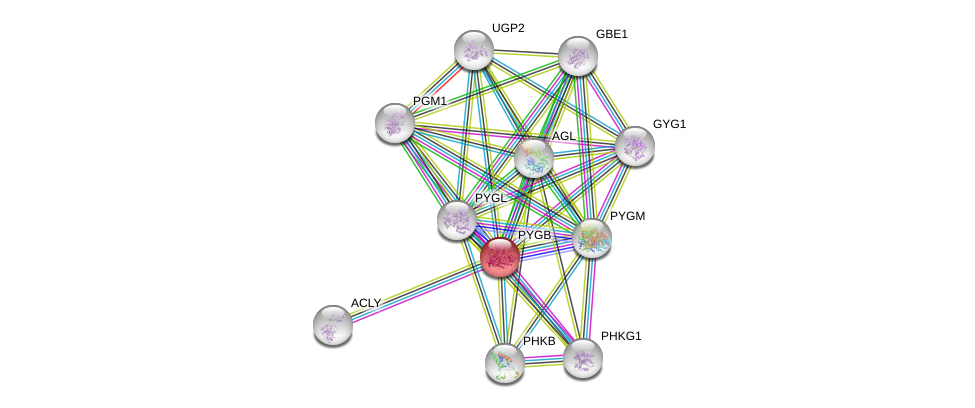
| Ensembl ID | Gene Symbol | Coverage | Identiy | Ortholog | Gene Symbol | Coverage | Identiy | Species |
|---|---|---|---|---|---|---|---|---|
| ENSG00000100994 | PYGB | 99 | 72.434 | FBgn0004507 | GlyP | 99 | 72.434 | Drosophila_melanogaster |
| ENSG00000100994 | PYGB | 99 | 81.883 | ENSG00000100504 | PYGL | 99 | 81.883 | Homo_sapiens |
| ENSG00000100994 | PYGB | 100 | 95.611 | ENSMUSG00000033059 | Pygb | 100 | 95.611 | Mus_musculus |
| ENSG00000100994 | PYGB | 99 | 83.671 | ENSMUSG00000032648 | Pygm | 99 | 83.671 | Mus_musculus |
| ENSG00000100994 | PYGB | 95 | 48.931 | YPR160W | 92 | 48.456 | Saccharomyces_cerevisiae |
| Go ID | Go_term | PubmedID | Evidence | Category |
|---|---|---|---|---|
| GO:0005515 | protein binding | 25416956.25910212. | IPI | Function |
| GO:0005576 | extracellular region | - | TAS | Component |
| GO:0005737 | cytoplasm | 21873635. | IBA | Component |
| GO:0005737 | cytoplasm | 10638593. | IDA | Component |
| GO:0005980 | glycogen catabolic process | 21873635. | IBA | Process |
| GO:0005980 | glycogen catabolic process | 3346228. | NAS | Process |
| GO:0008184 | glycogen phosphorylase activity | 21873635. | IBA | Function |
| GO:0008184 | glycogen phosphorylase activity | 3346228. | NAS | Function |
| GO:0016020 | membrane | 19946888. | HDA | Component |
| GO:0030170 | pyridoxal phosphate binding | 21873635. | IBA | Function |
| GO:0035578 | azurophil granule lumen | - | TAS | Component |
| GO:0043312 | neutrophil degranulation | - | TAS | Process |
| GO:0070062 | extracellular exosome | 23533145. | HDA | Component |
| GO:0102250 | linear malto-oligosaccharide phosphorylase activity | - | IEA | Function |
| GO:0102499 | SHG alpha-glucan phosphorylase activity | - | IEA | Function |
| PID | Title | Article | Time | Author | Doi |
|---|---|---|---|---|---|
| 30106110 |
| Cancer | Chr | Position | Mutation Type | dbSNP | Protein-change | Allele Freq | RBD |
|---|---|---|---|---|---|---|---|
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| DLBC | |||||||
| ESCA | |||||||
| ESCA | |||||||
| ESCA | |||||||
| ESCA | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| LGG | |||||||
| LGG | |||||||
| LGG | |||||||
| LGG | |||||||
| LIHC | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| MESO | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| PAAD | |||||||
| PAAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| READ | |||||||
| SARC | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCS |
| Cancer | Type | Freq | Q-value |
|---|---|---|---|
| LUAD |
| Cancer | P-value | Q-value |
|---|---|---|
| KIRC | ||
| STAD | ||
| MESO | ||
| ACC | ||
| LUSC | ||
| PAAD | ||
| BLCA | ||
| LIHC | ||
| LGG | ||
| CHOL | ||
| LUAD | ||
| UVM | ||
| OV |