
| Transcript ID | Name | Length | RefSeq ID | Protein ID | Length | RefSeq ID | UniportKB ID |
|---|---|---|---|---|---|---|---|
| ENST00000572019 | NUP88-203 | 580 | - | - | - (aa) | - | - |
| ENST00000572809 | NUP88-205 | 771 | - | ENSP00000459370 | 153 (aa) | - | I3L245 |
| ENST00000574855 | NUP88-209 | 434 | - | - | - (aa) | - | - |
| ENST00000575976 | NUP88-211 | 599 | - | - | - (aa) | - | - |
| ENST00000573584 | NUP88-207 | 2916 | XM_017024694 | ENSP00000458954 | 741 (aa) | XP_016880183 | Q99567 |
| ENST00000572290 | NUP88-204 | 615 | - | ENSP00000459979 | 128 (aa) | - | I3L2W3 |
| ENST00000573169 | NUP88-206 | 1195 | - | - | - (aa) | - | - |
| ENST00000576862 | NUP88-213 | 788 | - | - | - (aa) | - | - |
| ENST00000570937 | NUP88-202 | 361 | - | - | - (aa) | - | - |
| ENST00000576708 | NUP88-212 | 800 | - | ENSP00000461334 | 110 (aa) | - | I3L4K7 |
| ENST00000574087 | NUP88-208 | 548 | - | - | - (aa) | - | - |
| ENST00000574867 | NUP88-210 | 2081 | - | - | - (aa) | - | - |
| ENST00000225696 | NUP88-201 | 2194 | XM_011523893 | ENSP00000225696 | 696 (aa) | XP_011522195 | J3KMX1 |
| Pathway ID | Pathway Name | Source |
|---|---|---|
| hsa03013 | RNA transport | KEGG |
| ensgID | SNP | Chromosome | Position | SNP-risk | Trait | PubmedID | 95% CI | Or or BEAT | EFO ID |
|---|---|---|---|---|---|---|---|---|---|
| ENSG00000108559 | rs72634030 | 17 | 5369285 | A | Rheumatoid arthritis | 24390342 | [1.08-1.17] | 1.12 | EFO_0000685 |
| ENSG00000108559 | rs3026101 | 17 | 5377145 | C | Body mass index | 28892062 | [0.01-0.018] kg/m2 increase | 0.014 | EFO_0004340 |
| ENSG00000108559 | rs1000940 | 17 | 5379957 | A | Body mass index | 26426971 | unit decrease | 0.017490005 | EFO_0004340 |
| ENSG00000108559 | rs3026101 | 17 | 5377145 | T | Body mass index | 30108127 | [NR] unit decrease | 0.02 | EFO_0004340 |
| ENSG00000108559 | rs72634030 | 17 | 5369285 | A | Rheumatoid arthritis | 30423114 | [0.07-0.15] unit increase | 0.110343 | EFO_0000685 |
| ENSG00000108559 | rs10792 | 17 | 5384859 | ? | Body mass index | 30595370 | EFO_0004340 |
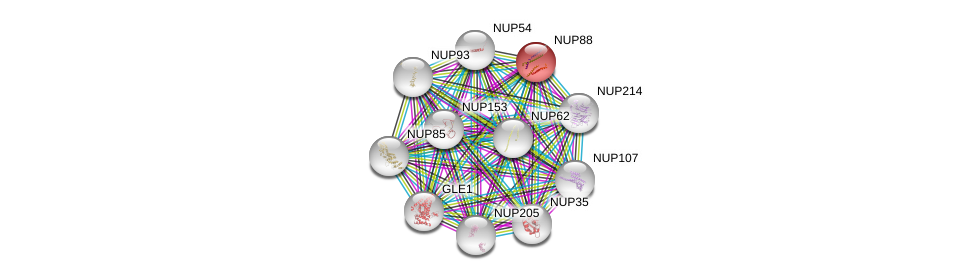
| Ensembl ID | Gene Symbol | Coverage | Identiy | Ortholog | Gene Symbol | Coverage | Identiy | Species |
|---|---|---|---|---|---|---|---|---|
| ENSG00000108559 | NUP88 | 100 | 89.906 | ENSMUSG00000040667 | Nup88 | 100 | 89.906 | Mus_musculus |
| Go ID | Go_term | PubmedID | Evidence | Category |
|---|---|---|---|---|
| GO:0000055 | ribosomal large subunit export from nucleus | 21873635. | IBA | Process |
| GO:0000056 | ribosomal small subunit export from nucleus | 21873635. | IBA | Process |
| GO:0000278 | mitotic cell cycle | - | IEA | Process |
| GO:0005215 | transporter activity | 9049309. | TAS | Function |
| GO:0005643 | nuclear pore | 21873635. | IBA | Component |
| GO:0005643 | nuclear pore | 23777819. | IDA | Component |
| GO:0005654 | nucleoplasm | - | IDA | Component |
| GO:0005829 | cytosol | - | TAS | Component |
| GO:0006110 | regulation of glycolytic process | - | TAS | Process |
| GO:0006406 | mRNA export from nucleus | 21873635. | IBA | Process |
| GO:0006406 | mRNA export from nucleus | - | TAS | Process |
| GO:0006409 | tRNA export from nucleus | - | TAS | Process |
| GO:0006606 | protein import into nucleus | 21873635. | IBA | Process |
| GO:0006611 | protein export from nucleus | 21873635. | IBA | Process |
| GO:0016032 | viral process | - | TAS | Process |
| GO:0016925 | protein sumoylation | - | TAS | Process |
| GO:0017056 | structural constituent of nuclear pore | - | IEA | Function |
| GO:0019083 | viral transcription | - | TAS | Process |
| GO:0043657 | host cell | - | IEA | Component |
| GO:0060964 | regulation of gene silencing by miRNA | - | TAS | Process |
| GO:0075733 | intracellular transport of virus | - | TAS | Process |
| GO:1900034 | regulation of cellular response to heat | - | TAS | Process |
| Cancer | Chr | Position | Mutation Type | dbSNP | Protein-change | Allele Freq | RBD |
|---|---|---|---|---|---|---|---|
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| ESCA | |||||||
| ESCA | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRP | |||||||
| KIRP | |||||||
| LAML | |||||||
| LGG | |||||||
| LGG | |||||||
| LGG | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| MESO | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| PAAD | |||||||
| PAAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| TGCT | |||||||
| THCA | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC |
| Cancer | Type | Freq | Q-value |
|---|---|---|---|
| DLBC | |||
| LAML | |||
| LIHC | |||
| THCA |
| Cancer | P-value | Q-value |
|---|---|---|
| THYM | ||
| SARC | ||
| ACC | ||
| PRAD | ||
| ESCA | ||
| PAAD | ||
| CESC | ||
| READ | ||
| KICH | ||
| UVM |