
| Protein ID | Domain | Pfam ID | E-value | Domain number | Total number |
|---|---|---|---|---|---|
| ENSP00000413812 | Ribonuc_2-5A | PF06479.12 | 3.8e-39 | 1 | 1 |
| ENSP00000256797 | Ribonuc_2-5A | PF06479.12 | 4.1e-39 | 1 | 1 |
| Transcript ID | Name | Length | RefSeq ID | Protein ID | Length | RefSeq ID | UniportKB ID |
|---|---|---|---|---|---|---|---|
| ENST00000566565 | ERN2-206 | 313 | - | - | - (aa) | - | - |
| ENST00000457008 | ERN2-202 | 3169 | - | ENSP00000413812 | 874 (aa) | - | E7ETG2 |
| ENST00000561478 | ERN2-203 | 412 | - | - | - (aa) | - | - |
| ENST00000256797 | ERN2-201 | 3549 | XM_011545708 | ENSP00000256797 | 926 (aa) | XP_011544010 | Q76MJ5 |
| ENST00000562458 | ERN2-204 | 759 | - | ENSP00000456866 | 23 (aa) | - | H3BSU1 |
| ENST00000569903 | ERN2-207 | 980 | - | ENSP00000457796 | 80 (aa) | - | H3BUU2 |
| ENST00000562562 | ERN2-205 | 2263 | XM_011545712 | ENSP00000457361 | 129 (aa) | XP_011544014 | H3BTW8 |
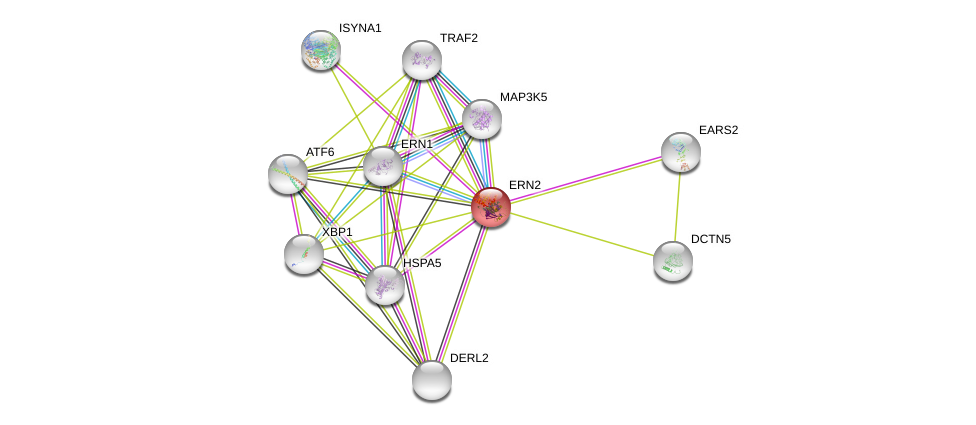
| Ensembl ID | Gene Symbol | Coverage | Identiy | Paralog | Gene Symbol | Coverage | Identiy |
|---|---|---|---|---|---|---|---|
| ENSG00000134398 | ERN2 | 94 | 65.517 | ENSG00000178607 | ERN1 | 87 | 63.387 |
| Ensembl ID | Gene Symbol | Coverage | Identiy | Ortholog | Gene Symbol | Coverage | Identiy | Species |
|---|---|---|---|---|---|---|---|---|
| ENSG00000134398 | ERN2 | 97 | 87.653 | ENSAMEG00000014205 | ERN2 | 100 | 86.222 | Ailuropoda_melanoleuca |
| ENSG00000134398 | ERN2 | 92 | 64.944 | ENSAPLG00000010981 | ERN2 | 96 | 55.449 | Anas_platyrhynchos |
| ENSG00000134398 | ERN2 | 96 | 56.503 | ENSACAG00000012164 | ERN2 | 97 | 56.813 | Anolis_carolinensis |
| ENSG00000134398 | ERN2 | 100 | 95.680 | ENSANAG00000016991 | ERN2 | 100 | 95.680 | Aotus_nancymaae |
| ENSG00000134398 | ERN2 | 99 | 84.096 | ENSBTAG00000014456 | ERN2 | 99 | 84.211 | Bos_taurus |
| ENSG00000134398 | ERN2 | 100 | 94.060 | ENSCJAG00000020632 | ERN2 | 100 | 94.060 | Callithrix_jacchus |
| ENSG00000134398 | ERN2 | 97 | 85.033 | ENSCAFG00000017643 | ERN2 | 99 | 84.416 | Canis_familiaris |
| ENSG00000134398 | ERN2 | 97 | 82.262 | ENSCAFG00020021342 | ERN2 | 99 | 81.710 | Canis_lupus_dingo |
| ENSG00000134398 | ERN2 | 99 | 85.005 | ENSCHIG00000022191 | ERN2 | 99 | 85.545 | Capra_hircus |
| ENSG00000134398 | ERN2 | 99 | 89.480 | ENSTSYG00000001334 | ERN2 | 99 | 89.942 | Carlito_syrichta |
| ENSG00000134398 | ERN2 | 91 | 74.125 | ENSCAPG00000005936 | ERN2 | 94 | 84.112 | Cavia_aperea |
| ENSG00000134398 | ERN2 | 99 | 83.134 | ENSCPOG00000005916 | ERN2 | 99 | 83.134 | Cavia_porcellus |
| ENSG00000134398 | ERN2 | 100 | 94.737 | ENSCCAG00000035789 | ERN2 | 100 | 94.924 | Cebus_capucinus |
| ENSG00000134398 | ERN2 | 100 | 96.652 | ENSCATG00000040324 | ERN2 | 100 | 96.652 | Cercocebus_atys |
| ENSG00000134398 | ERN2 | 96 | 82.735 | ENSCLAG00000014120 | ERN2 | 100 | 82.735 | Chinchilla_lanigera |
| ENSG00000134398 | ERN2 | 100 | 95.910 | ENSCSAG00000006703 | ERN2 | 100 | 95.910 | Chlorocebus_sabaeus |
| ENSG00000134398 | ERN2 | 52 | 77.404 | ENSCHOG00000010014 | - | 81 | 77.404 | Choloepus_hoffmanni |
| ENSG00000134398 | ERN2 | 89 | 71.679 | ENSCPBG00000002165 | ERN2 | 94 | 58.849 | Chrysemys_picta_bellii |
| ENSG00000134398 | ERN2 | 100 | 96.436 | ENSCANG00000015296 | ERN2 | 100 | 96.436 | Colobus_angolensis_palliatus |
| ENSG00000134398 | ERN2 | 96 | 84.086 | ENSCGRG00001019098 | Ern2 | 100 | 84.086 | Cricetulus_griseus_chok1gshd |
| ENSG00000134398 | ERN2 | 97 | 80.894 | ENSDNOG00000017619 | ERN2 | 97 | 80.894 | Dasypus_novemcinctus |
| ENSG00000134398 | ERN2 | 99 | 82.890 | ENSDORG00000001246 | Ern2 | 97 | 84.099 | Dipodomys_ordii |
| ENSG00000134398 | ERN2 | 92 | 85.277 | ENSETEG00000019530 | ERN2 | 98 | 85.277 | Echinops_telfairi |
| ENSG00000134398 | ERN2 | 50 | 58.581 | ENSEBUG00000003994 | - | 90 | 54.183 | Eptatretus_burgeri |
| ENSG00000134398 | ERN2 | 99 | 85.022 | ENSEASG00005017645 | ERN2 | 99 | 84.682 | Equus_asinus_asinus |
| ENSG00000134398 | ERN2 | 99 | 87.055 | ENSECAG00000017742 | ERN2 | 99 | 86.717 | Equus_caballus |
| ENSG00000134398 | ERN2 | 89 | 76.387 | ENSEEUG00000005382 | ERN2 | 86 | 76.387 | Erinaceus_europaeus |
| ENSG00000134398 | ERN2 | 99 | 84.800 | ENSFCAG00000012945 | ERN2 | 99 | 85.371 | Felis_catus |
| ENSG00000134398 | ERN2 | 91 | 71.223 | ENSFALG00000003790 | ERN2 | 84 | 71.223 | Ficedula_albicollis |
| ENSG00000134398 | ERN2 | 98 | 80.592 | ENSFDAG00000006000 | ERN2 | 98 | 82.426 | Fukomys_damarensis |
| ENSG00000134398 | ERN2 | 89 | 71.178 | ENSGAGG00000002490 | ERN2 | 89 | 63.025 | Gopherus_agassizii |
| ENSG00000134398 | ERN2 | 100 | 98.812 | ENSGGOG00000012853 | ERN2 | 100 | 98.812 | Gorilla_gorilla |
| ENSG00000134398 | ERN2 | 99 | 84.855 | ENSHGLG00000012125 | ERN2 | 97 | 85.318 | Heterocephalus_glaber_female |
| ENSG00000134398 | ERN2 | 96 | 85.442 | ENSHGLG00100009410 | ERN2 | 96 | 85.442 | Heterocephalus_glaber_male |
| ENSG00000134398 | ERN2 | 97 | 86.272 | ENSSTOG00000012411 | ERN2 | 99 | 85.854 | Ictidomys_tridecemlineatus |
| ENSG00000134398 | ERN2 | 96 | 84.400 | ENSJJAG00000015169 | Ern2 | 96 | 84.400 | Jaculus_jaculus |
| ENSG00000134398 | ERN2 | 97 | 87.584 | ENSLAFG00000005429 | ERN2 | 99 | 86.457 | Loxodonta_africana |
| ENSG00000134398 | ERN2 | 83 | 83.377 | ENSMFAG00000038350 | ERN2 | 92 | 83.377 | Macaca_fascicularis |
| ENSG00000134398 | ERN2 | 100 | 96.544 | ENSMMUG00000013806 | ERN2 | 100 | 96.453 | Macaca_mulatta |
| ENSG00000134398 | ERN2 | 100 | 96.652 | ENSMNEG00000034382 | ERN2 | 100 | 96.652 | Macaca_nemestrina |
| ENSG00000134398 | ERN2 | 89 | 96.590 | ENSMLEG00000040622 | ERN2 | 100 | 96.590 | Mandrillus_leucophaeus |
| ENSG00000134398 | ERN2 | 98 | 67.603 | ENSMGAG00000006523 | ERN2 | 99 | 59.201 | Meleagris_gallopavo |
| ENSG00000134398 | ERN2 | 87 | 83.704 | ENSMAUG00000013187 | Ern2 | 91 | 81.420 | Mesocricetus_auratus |
| ENSG00000134398 | ERN2 | 99 | 86.270 | ENSMICG00000007841 | ERN2 | 99 | 86.054 | Microcebus_murinus |
| ENSG00000134398 | ERN2 | 99 | 82.826 | ENSMOCG00000000736 | Ern2 | 98 | 82.826 | Microtus_ochrogaster |
| ENSG00000134398 | ERN2 | 96 | 66.882 | ENSMODG00000006717 | ERN2 | 97 | 66.882 | Monodelphis_domestica |
| ENSG00000134398 | ERN2 | 99 | 81.828 | MGP_CAROLIEiJ_G0030420 | Ern2 | 99 | 82.995 | Mus_caroli |
| ENSG00000134398 | ERN2 | 99 | 81.610 | ENSMUSG00000030866 | Ern2 | 99 | 81.610 | Mus_musculus |
| ENSG00000134398 | ERN2 | 99 | 81.937 | MGP_PahariEiJ_G0013630 | Ern2 | 99 | 83.333 | Mus_pahari |
| ENSG00000134398 | ERN2 | 99 | 81.828 | MGP_SPRETEiJ_G0031534 | Ern2 | 99 | 82.995 | Mus_spretus |
| ENSG00000134398 | ERN2 | 99 | 85.097 | ENSMPUG00000012189 | ERN2 | 99 | 85.653 | Mustela_putorius_furo |
| ENSG00000134398 | ERN2 | 89 | 76.428 | ENSMLUG00000012167 | ERN2 | 99 | 76.521 | Myotis_lucifugus |
| ENSG00000134398 | ERN2 | 98 | 83.942 | ENSNGAG00000021537 | Ern2 | 97 | 84.129 | Nannospalax_galili |
| ENSG00000134398 | ERN2 | 100 | 94.384 | ENSNLEG00000011950 | ERN2 | 100 | 94.384 | Nomascus_leucogenys |
| ENSG00000134398 | ERN2 | 95 | 63.949 | ENSOANG00000007667 | - | 99 | 63.949 | Ornithorhynchus_anatinus |
| ENSG00000134398 | ERN2 | 99 | 81.885 | ENSOCUG00000008483 | ERN2 | 99 | 82.408 | Oryctolagus_cuniculus |
| ENSG00000134398 | ERN2 | 99 | 86.659 | ENSOGAG00000005577 | ERN2 | 97 | 87.946 | Otolemur_garnettii |
| ENSG00000134398 | ERN2 | 97 | 84.633 | ENSOARG00000017432 | ERN2 | 99 | 84.633 | Ovis_aries |
| ENSG00000134398 | ERN2 | 100 | 99.136 | ENSPPAG00000037128 | ERN2 | 100 | 99.136 | Pan_paniscus |
| ENSG00000134398 | ERN2 | 99 | 84.898 | ENSPPRG00000001852 | ERN2 | 99 | 85.437 | Panthera_pardus |
| ENSG00000134398 | ERN2 | 97 | 83.501 | ENSPTIG00000016188 | ERN2 | 99 | 82.435 | Panthera_tigris_altaica |
| ENSG00000134398 | ERN2 | 100 | 99.244 | ENSPTRG00000007893 | ERN2 | 100 | 99.244 | Pan_troglodytes |
| ENSG00000134398 | ERN2 | 100 | 96.436 | ENSPANG00000023632 | ERN2 | 100 | 96.436 | Papio_anubis |
| ENSG00000134398 | ERN2 | 96 | 83.708 | ENSPEMG00000012773 | Ern2 | 97 | 83.557 | Peromyscus_maniculatus_bairdii |
| ENSG00000134398 | ERN2 | 94 | 67.841 | ENSPCIG00000026605 | ERN2 | 95 | 67.841 | Phascolarctos_cinereus |
| ENSG00000134398 | ERN2 | 100 | 97.732 | ENSPPYG00000007196 | ERN2 | 95 | 97.732 | Pongo_abelii |
| ENSG00000134398 | ERN2 | 99 | 82.792 | ENSPCAG00000016200 | ERN2 | 93 | 83.667 | Procavia_capensis |
| ENSG00000134398 | ERN2 | 97 | 87.678 | ENSPCOG00000017614 | ERN2 | 98 | 87.618 | Propithecus_coquereli |
| ENSG00000134398 | ERN2 | 99 | 78.162 | ENSPVAG00000008445 | ERN2 | 95 | 78.595 | Pteropus_vampyrus |
| ENSG00000134398 | ERN2 | 99 | 80.978 | ENSRNOG00000018974 | Ern2 | 98 | 80.978 | Rattus_norvegicus |
| ENSG00000134398 | ERN2 | 100 | 96.682 | ENSRBIG00000034872 | ERN2 | 100 | 96.682 | Rhinopithecus_bieti |
| ENSG00000134398 | ERN2 | 100 | 96.652 | ENSRROG00000008752 | ERN2 | 100 | 96.652 | Rhinopithecus_roxellana |
| ENSG00000134398 | ERN2 | 85 | 88.732 | ENSSBOG00000027251 | ERN2 | 96 | 88.732 | Saimiri_boliviensis_boliviensis |
| ENSG00000134398 | ERN2 | 94 | 68.274 | ENSSHAG00000005538 | ERN2 | 98 | 68.274 | Sarcophilus_harrisii |
| ENSG00000134398 | ERN2 | 78 | 73.984 | ENSSPUG00000010312 | ERN2 | 91 | 60.938 | Sphenodon_punctatus |
| ENSG00000134398 | ERN2 | 99 | 85.297 | ENSSSCG00000040637 | ERN2 | 93 | 85.405 | Sus_scrofa |
| ENSG00000134398 | ERN2 | 86 | 72.682 | ENSTGUG00000006039 | ERN2 | 99 | 57.811 | Taeniopygia_guttata |
| ENSG00000134398 | ERN2 | 97 | 87.025 | ENSTBEG00000000414 | ERN2 | 99 | 87.472 | Tupaia_belangeri |
| ENSG00000134398 | ERN2 | 92 | 81.935 | ENSTTRG00000005264 | ERN2 | 95 | 81.702 | Tursiops_truncatus |
| ENSG00000134398 | ERN2 | 97 | 84.316 | ENSUAMG00000021038 | ERN2 | 100 | 82.992 | Ursus_americanus |
| ENSG00000134398 | ERN2 | 97 | 81.646 | ENSUMAG00000008840 | ERN2 | 97 | 81.646 | Ursus_maritimus |
| ENSG00000134398 | ERN2 | 99 | 83.459 | ENSVVUG00000005617 | ERN2 | 99 | 84.017 | Vulpes_vulpes |
| Go ID | Go_term | PubmedID | Evidence | Category |
|---|---|---|---|---|
| GO:0000287 | magnesium ion binding | 11175748. | IDA | Function |
| GO:0004519 | endonuclease activity | 11175748. | IDA | Function |
| GO:0004521 | endoribonuclease activity | 21873635. | IBA | Function |
| GO:0004672 | protein kinase activity | 21873635. | IBA | Function |
| GO:0004674 | protein serine/threonine kinase activity | 21873635. | IBA | Function |
| GO:0004674 | protein serine/threonine kinase activity | 11175748. | IDA | Function |
| GO:0005524 | ATP binding | 11175748. | IDA | Function |
| GO:0005789 | endoplasmic reticulum membrane | 21873635. | IBA | Component |
| GO:0006397 | mRNA processing | - | IEA | Process |
| GO:0006468 | protein phosphorylation | 11175748. | IDA | Process |
| GO:0007050 | cell cycle arrest | - | IEA | Process |
| GO:0007257 | activation of JUN kinase activity | - | IEA | Process |
| GO:0016021 | integral component of membrane | - | IEA | Component |
| GO:0016075 | rRNA catabolic process | 11175748. | IDA | Process |
| GO:0030263 | apoptotic chromosome condensation | 11175748. | IMP | Process |
| GO:0030968 | endoplasmic reticulum unfolded protein response | 21873635. | IBA | Process |
| GO:0034976 | response to endoplasmic reticulum stress | 22013210. | TAS | Process |
| GO:0036498 | IRE1-mediated unfolded protein response | 21873635. | IBA | Process |
| GO:0045892 | negative regulation of transcription, DNA-templated | 11175748. | IMP | Process |
| GO:0051082 | unfolded protein binding | 21873635. | IBA | Function |
| GO:0070059 | intrinsic apoptotic signaling pathway in response to endoplasmic reticulum stress | 21873635. | IBA | Process |
| GO:0090502 | RNA phosphodiester bond hydrolysis, endonucleolytic | - | IEA | Process |
| GO:1990604 | IRE1-TRAF2-ASK1 complex | 21873635. | IBA | Component |
| GO:1990604 | IRE1-TRAF2-ASK1 complex | - | ISS | Component |
| Cancer | Chr | Position | Mutation Type | dbSNP | Protein-change | Allele Freq | RBD |
|---|---|---|---|---|---|---|---|
| ACC | |||||||
| ACC | |||||||
| ACC | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| ESCA | |||||||
| ESCA | |||||||
| ESCA | |||||||
| ESCA | |||||||
| ESCA | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| KIRC | |||||||
| KIRP | |||||||
| LGG | |||||||
| LGG | |||||||
| LGG | |||||||
| LGG | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| PAAD | |||||||
| PAAD | |||||||
| PAAD | |||||||
| PAAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| SARC | |||||||
| SARC | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| THYM | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCS |
| Cancer | Type | Freq | Q-value |
|---|---|---|---|
| CHOL | |||
| GBM |
| Cancer | P-value | Q-value |
|---|---|---|
| HNSC | ||
| SKCM | ||
| PAAD | ||
| BLCA | ||
| CESC | ||
| LAML | ||
| LIHC | ||
| DLBC | ||
| LGG | ||
| UVM |