
| Protein ID | Domain | Pfam ID | E-value | Domain number | Total number |
|---|---|---|---|---|---|
| ENSP00000319829 | zf-C2H2 | PF00096.26 | 7.7e-14 | 1 | 2 |
| ENSP00000319829 | zf-C2H2 | PF00096.26 | 7.7e-14 | 2 | 2 |
| ENSP00000336620 | zf-C2H2 | PF00096.26 | 7.7e-14 | 1 | 2 |
| ENSP00000336620 | zf-C2H2 | PF00096.26 | 7.7e-14 | 2 | 2 |
| ENSP00000398821 | zf-C2H2 | PF00096.26 | 2.1e-09 | 1 | 1 |
| ENSP00000319829 | zf-met | PF12874.7 | 1.6e-05 | 1 | 1 |
| ENSP00000336620 | zf-met | PF12874.7 | 1.6e-05 | 1 | 1 |
| PID | Title | Method | Time | Author | Doi |
|---|---|---|---|---|---|
| 30607034 |
| Transcript ID | Name | Length | RefSeq ID | Protein ID | Length | RefSeq ID | UniportKB ID |
|---|---|---|---|---|---|---|---|
| ENST00000426871 | ZNF687-203 | 3277 | - | ENSP00000398821 | 706 (aa) | - | H0Y5I5 |
| ENST00000459919 | ZNF687-207 | 840 | - | - | - (aa) | - | - |
| ENST00000443959 | ZNF687-205 | 893 | - | ENSP00000395261 | 147 (aa) | - | A2A3Q2 |
| ENST00000324048 | ZNF687-201 | 5378 | - | ENSP00000319829 | 1237 (aa) | - | Q8N1G0 |
| ENST00000449313 | ZNF687-206 | 4185 | - | ENSP00000415286 | 785 (aa) | - | F8WCX2 |
| ENST00000336715 | ZNF687-202 | 4831 | XM_005245366 | ENSP00000336620 | 1237 (aa) | XP_005245423 | Q8N1G0 |
| ENST00000436614 | ZNF687-204 | 766 | - | ENSP00000416942 | 99 (aa) | - | A6PVV7 |
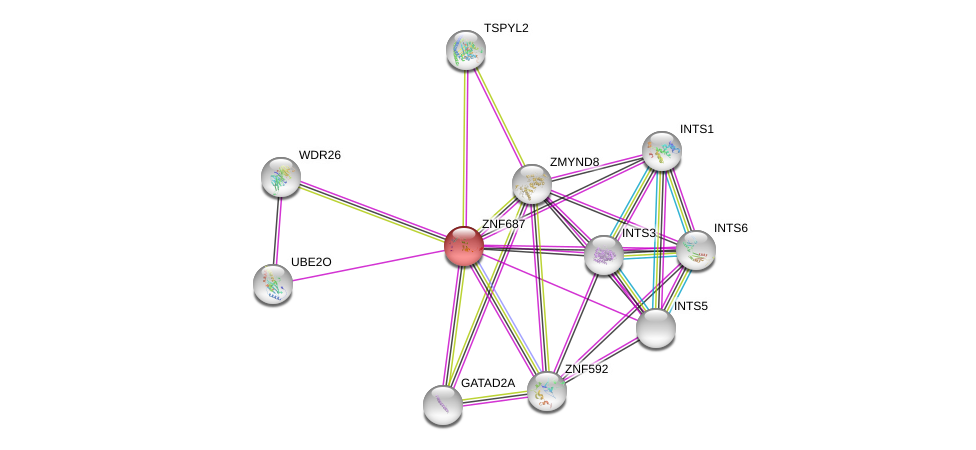
| Ensembl ID | Gene Symbol | Coverage | Identiy | Ortholog | Gene Symbol | Coverage | Identiy | Species |
|---|---|---|---|---|---|---|---|---|
| ENSG00000143373 | ZNF687 | 100 | 88.468 | ENSAMEG00000000725 | ZNF687 | 100 | 88.306 | Ailuropoda_melanoleuca |
| ENSG00000143373 | ZNF687 | 100 | 95.557 | ENSANAG00000025948 | ZNF687 | 100 | 95.396 | Aotus_nancymaae |
| ENSG00000143373 | ZNF687 | 100 | 90.638 | ENSBTAG00000007318 | ZNF687 | 100 | 90.638 | Bos_taurus |
| ENSG00000143373 | ZNF687 | 100 | 94.992 | ENSCJAG00000033395 | ZNF687 | 100 | 94.830 | Callithrix_jacchus |
| ENSG00000143373 | ZNF687 | 100 | 88.629 | ENSCAFG00000012605 | ZNF687 | 100 | 88.468 | Canis_familiaris |
| ENSG00000143373 | ZNF687 | 100 | 88.629 | ENSCAFG00020012909 | ZNF687 | 100 | 88.468 | Canis_lupus_dingo |
| ENSG00000143373 | ZNF687 | 100 | 91.935 | ENSTSYG00000033410 | ZNF687 | 100 | 91.774 | Carlito_syrichta |
| ENSG00000143373 | ZNF687 | 100 | 84.045 | ENSCAPG00000017697 | ZNF687 | 100 | 83.642 | Cavia_aperea |
| ENSG00000143373 | ZNF687 | 100 | 85.415 | ENSCPOG00000010631 | ZNF687 | 100 | 85.012 | Cavia_porcellus |
| ENSG00000143373 | ZNF687 | 100 | 95.153 | ENSCCAG00000036501 | ZNF687 | 100 | 94.992 | Cebus_capucinus |
| ENSG00000143373 | ZNF687 | 100 | 97.979 | ENSCATG00000000360 | ZNF687 | 100 | 97.979 | Cercocebus_atys |
| ENSG00000143373 | ZNF687 | 100 | 86.463 | ENSCLAG00000000787 | ZNF687 | 100 | 86.301 | Chinchilla_lanigera |
| ENSG00000143373 | ZNF687 | 68 | 97.495 | ENSCSAG00000001797 | ZNF687 | 100 | 98.825 | Chlorocebus_sabaeus |
| ENSG00000143373 | ZNF687 | 100 | 97.332 | ENSCANG00000037537 | ZNF687 | 100 | 97.332 | Colobus_angolensis_palliatus |
| ENSG00000143373 | ZNF687 | 100 | 84.875 | ENSCGRG00001011676 | Zfp687 | 100 | 84.232 | Cricetulus_griseus_chok1gshd |
| ENSG00000143373 | ZNF687 | 100 | 84.875 | ENSCGRG00000010527 | Zfp687 | 100 | 84.232 | Cricetulus_griseus_crigri |
| ENSG00000143373 | ZNF687 | 100 | 91.855 | ENSDNOG00000046703 | ZNF687 | 100 | 91.694 | Dasypus_novemcinctus |
| ENSG00000143373 | ZNF687 | 89 | 95.231 | ENSEASG00005002145 | ZNF687 | 100 | 91.765 | Equus_asinus_asinus |
| ENSG00000143373 | ZNF687 | 100 | 90.726 | ENSECAG00000006072 | ZNF687 | 100 | 90.000 | Equus_caballus |
| ENSG00000143373 | ZNF687 | 100 | 87.823 | ENSFCAG00000005566 | ZNF687 | 100 | 86.452 | Felis_catus |
| ENSG00000143373 | ZNF687 | 100 | 99.030 | ENSGGOG00000015144 | ZNF687 | 100 | 99.030 | Gorilla_gorilla |
| ENSG00000143373 | ZNF687 | 100 | 86.774 | ENSHGLG00000009054 | ZNF687 | 100 | 87.016 | Heterocephalus_glaber_female |
| ENSG00000143373 | ZNF687 | 100 | 90.403 | ENSSTOG00000023367 | ZNF687 | 100 | 90.242 | Ictidomys_tridecemlineatus |
| ENSG00000143373 | ZNF687 | 100 | 91.935 | ENSLAFG00000030899 | ZNF687 | 100 | 91.774 | Loxodonta_africana |
| ENSG00000143373 | ZNF687 | 100 | 97.575 | ENSMFAG00000034438 | ZNF687 | 100 | 97.575 | Macaca_fascicularis |
| ENSG00000143373 | ZNF687 | 100 | 97.736 | ENSMMUG00000013166 | ZNF687 | 100 | 97.736 | Macaca_mulatta |
| ENSG00000143373 | ZNF687 | 100 | 97.656 | ENSMNEG00000034210 | ZNF687 | 100 | 97.656 | Macaca_nemestrina |
| ENSG00000143373 | ZNF687 | 100 | 97.979 | ENSMLEG00000024831 | ZNF687 | 100 | 97.979 | Mandrillus_leucophaeus |
| ENSG00000143373 | ZNF687 | 100 | 83.977 | ENSMAUG00000004550 | Zfp687 | 100 | 83.333 | Mesocricetus_auratus |
| ENSG00000143373 | ZNF687 | 100 | 88.245 | ENSMICG00000016128 | ZNF687 | 100 | 88.084 | Microcebus_murinus |
| ENSG00000143373 | ZNF687 | 100 | 86.484 | MGP_CAROLIEiJ_G0025323 | Zfp687 | 100 | 85.749 | Mus_caroli |
| ENSG00000143373 | ZNF687 | 100 | 85.921 | ENSMUSG00000019338 | Zfp687 | 100 | 85.185 | Mus_musculus |
| ENSG00000143373 | ZNF687 | 100 | 85.173 | MGP_PahariEiJ_G0026767 | Zfp687 | 100 | 85.093 | Mus_pahari |
| ENSG00000143373 | ZNF687 | 100 | 86.082 | MGP_SPRETEiJ_G0026267 | Zfp687 | 100 | 85.346 | Mus_spretus |
| ENSG00000143373 | ZNF687 | 100 | 88.710 | ENSMPUG00000003797 | ZNF687 | 100 | 88.548 | Mustela_putorius_furo |
| ENSG00000143373 | ZNF687 | 100 | 86.301 | ENSNGAG00000017739 | Zfp687 | 100 | 85.576 | Nannospalax_galili |
| ENSG00000143373 | ZNF687 | 100 | 98.060 | ENSNLEG00000010154 | ZNF687 | 100 | 98.060 | Nomascus_leucogenys |
| ENSG00000143373 | ZNF687 | 100 | 86.371 | ENSODEG00000010274 | ZNF687 | 100 | 86.210 | Octodon_degus |
| ENSG00000143373 | ZNF687 | 100 | 89.326 | ENSOCUG00000027462 | ZNF687 | 100 | 87.881 | Oryctolagus_cuniculus |
| ENSG00000143373 | ZNF687 | 100 | 86.855 | ENSOGAG00000032200 | ZNF687 | 100 | 85.161 | Otolemur_garnettii |
| ENSG00000143373 | ZNF687 | 100 | 99.596 | ENSPPAG00000027876 | ZNF687 | 100 | 99.596 | Pan_paniscus |
| ENSG00000143373 | ZNF687 | 100 | 88.145 | ENSPPRG00000003190 | ZNF687 | 100 | 86.774 | Panthera_pardus |
| ENSG00000143373 | ZNF687 | 100 | 87.440 | ENSPTIG00000007625 | ZNF687 | 100 | 86.071 | Panthera_tigris_altaica |
| ENSG00000143373 | ZNF687 | 100 | 99.677 | ENSPTRG00000001284 | ZNF687 | 100 | 99.677 | Pan_troglodytes |
| ENSG00000143373 | ZNF687 | 100 | 97.979 | ENSPANG00000003115 | ZNF687 | 100 | 97.979 | Papio_anubis |
| ENSG00000143373 | ZNF687 | 100 | 85.048 | ENSPEMG00000016093 | Zfp687 | 100 | 85.209 | Peromyscus_maniculatus_bairdii |
| ENSG00000143373 | ZNF687 | 100 | 98.383 | ENSPPYG00000000869 | ZNF687 | 100 | 98.383 | Pongo_abelii |
| ENSG00000143373 | ZNF687 | 100 | 88.790 | ENSPCAG00000010893 | ZNF687 | 100 | 87.742 | Procavia_capensis |
| ENSG00000143373 | ZNF687 | 100 | 89.032 | ENSPCOG00000024950 | ZNF687 | 100 | 88.871 | Propithecus_coquereli |
| ENSG00000143373 | ZNF687 | 86 | 86.929 | ENSPVAG00000011674 | ZNF687 | 86 | 86.929 | Pteropus_vampyrus |
| ENSG00000143373 | ZNF687 | 100 | 85.829 | ENSRNOG00000021026 | Zfp687 | 100 | 84.863 | Rattus_norvegicus |
| ENSG00000143373 | ZNF687 | 100 | 97.575 | ENSRBIG00000028192 | ZNF687 | 100 | 97.575 | Rhinopithecus_bieti |
| ENSG00000143373 | ZNF687 | 100 | 97.736 | ENSRROG00000037226 | ZNF687 | 100 | 97.736 | Rhinopithecus_roxellana |
| ENSG00000143373 | ZNF687 | 100 | 95.153 | ENSSBOG00000036275 | ZNF687 | 100 | 94.992 | Saimiri_boliviensis_boliviensis |
| ENSG00000143373 | ZNF687 | 100 | 90.499 | ENSSSCG00000006627 | ZNF687 | 100 | 90.338 | Sus_scrofa |
| ENSG00000143373 | ZNF687 | 100 | 90.806 | ENSTTRG00000007020 | ZNF687 | 100 | 90.645 | Tursiops_truncatus |
| ENSG00000143373 | ZNF687 | 100 | 89.435 | ENSUAMG00000027445 | ZNF687 | 100 | 89.435 | Ursus_americanus |
| ENSG00000143373 | ZNF687 | 100 | 88.468 | ENSVVUG00000012829 | ZNF687 | 100 | 88.306 | Vulpes_vulpes |
| ENSG00000143373 | ZNF687 | 89 | 63.206 | ENSXETG00000010001 | znf687 | 59 | 60.669 | Xenopus_tropicalis |
| Go ID | Go_term | PubmedID | Evidence | Category |
|---|---|---|---|---|
| GO:0000981 | DNA-binding transcription factor activity, RNA polymerase II-specific | - | ISA | Function |
| GO:0003677 | DNA binding | - | IEA | Function |
| GO:0005515 | protein binding | 26496610. | IPI | Function |
| GO:0005654 | nucleoplasm | - | IDA | Component |
| GO:0005829 | cytosol | - | IDA | Component |
| GO:0006357 | regulation of transcription by RNA polymerase II | - | IEA | Process |
| GO:0046872 | metal ion binding | - | IEA | Function |
| Cancer | Chr | Position | Mutation Type | dbSNP | Protein-change | Allele Freq | RBD |
|---|---|---|---|---|---|---|---|
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| ESCA | |||||||
| ESCA | |||||||
| ESCA | |||||||
| ESCA | |||||||
| ESCA | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| LAML | |||||||
| LAML | |||||||
| LGG | |||||||
| LGG | |||||||
| LGG | |||||||
| LGG | |||||||
| LGG | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| MESO | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| PAAD | |||||||
| PAAD | |||||||
| PAAD | |||||||
| PCPG | |||||||
| PRAD | |||||||
| PRAD | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| SARC | |||||||
| SARC | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| THCA | |||||||
| THYM | |||||||
| THYM | |||||||
| THYM | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCS |
| Cancer | Type | Freq | Q-value |
|---|---|---|---|
| ACC | |||
| CESC | |||
| DLBC | |||
| ESCA | |||
| KIRC | |||
| LUSC | |||
| PAAD | |||
| PCPG | |||
| READ | |||
| SARC | |||
| SKCM | |||
| TGCT |
| Cancer | P-value | Q-value |
|---|---|---|
| KIRC | ||
| ACC | ||
| SKCM | ||
| ESCA | ||
| KIRP | ||
| LAML | ||
| UCEC | ||
| LIHC | ||
| LGG | ||
| UVM |