
| PID | Title | Article | Time | Author | Doi |
|---|---|---|---|---|---|
| 10385389 |
| Transcript ID | Name | Length | RefSeq ID | Protein ID | Length | RefSeq ID | UniportKB ID |
|---|---|---|---|---|---|---|---|
| ENST00000360270 | MSN-201 | 3944 | XM_005262269 | ENSP00000353408 | 577 (aa) | XP_005262326 | P26038 |
| ENST00000447323 | MSN-204 | 382 | - | - | - (aa) | - | - |
| ENST00000609205 | MSN-205 | 1761 | - | - | - (aa) | - | - |
| ENST00000439474 | MSN-203 | 539 | - | - | - (aa) | - | - |
| ENST00000609672 | MSN-206 | 568 | - | ENSP00000477441 | 103 (aa) | - | V9GZ54 |
| ENST00000429601 | MSN-202 | 487 | - | - | - (aa) | - | - |
| ensgID | SNP | Chromosome | Position | SNP-risk | Trait | PubmedID | 95% CI | Or or BEAT | EFO ID |
|---|---|---|---|---|---|---|---|---|---|
| ENSG00000147065 | rs143755874 | X | 65639521 | A | Male-pattern baldness | 28196072 | NR unit decrease | 0.1862 | Orphanet_79364 |
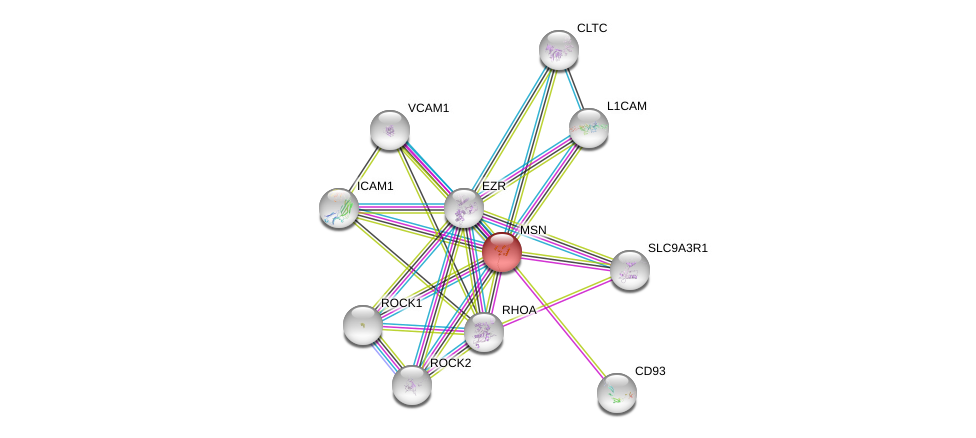
| Ensembl ID | Gene Symbol | Coverage | Identiy | Ortholog | Gene Symbol | Coverage | Identiy | Species |
|---|---|---|---|---|---|---|---|---|
| ENSG00000147065 | MSN | 100 | 88.350 | ENSG00000137710 | RDX | 100 | 89.655 | Homo_sapiens |
| ENSG00000147065 | MSN | 100 | 86.408 | ENSG00000092820 | EZR | 100 | 73.038 | Homo_sapiens |
| ENSG00000147065 | MSN | 100 | 99.029 | ENSMUSG00000031207 | Msn | 100 | 98.614 | Mus_musculus |
| ENSG00000147065 | MSN | 100 | 88.350 | ENSMUSG00000032050 | Rdx | 93 | 89.532 | Mus_musculus |
| ENSG00000147065 | MSN | 100 | 85.437 | ENSMUSG00000052397 | Ezr | 100 | 72.572 | Mus_musculus |
| Go ID | Go_term | PubmedID | Evidence | Category |
|---|---|---|---|---|
| GO:0001771 | immunological synapse formation | 27405666. | IDA | Process |
| GO:0001931 | uropod | - | IEA | Component |
| GO:0003725 | double-stranded RNA binding | 21266579. | IDA | Function |
| GO:0003779 | actin binding | - | IEA | Function |
| GO:0005102 | signaling receptor binding | 15819698. | IPI | Function |
| GO:0005200 | structural constituent of cytoskeleton | 1924289. | TAS | Function |
| GO:0005515 | protein binding | 7844168.9430655.10806479.15020681.16938849.17046996.18586956.20642453.21044950.21148287.22363216.22952686.23241358.23855374.24351927.24695735.24794857.24904275.25009464.26824392.27357661. | IPI | Function |
| GO:0005615 | extracellular space | 16502470. | HDA | Component |
| GO:0005634 | nucleus | 16791210. | HDA | Component |
| GO:0005737 | cytoplasm | 16791210. | HDA | Component |
| GO:0005829 | cytosol | - | IDA | Component |
| GO:0005829 | cytosol | - | TAS | Component |
| GO:0005856 | cytoskeleton | 16130169. | TAS | Component |
| GO:0005886 | plasma membrane | - | IDA | Component |
| GO:0005902 | microvillus | 12082081. | IDA | Component |
| GO:0005925 | focal adhesion | 21423176. | HDA | Component |
| GO:0005925 | focal adhesion | 21282464. | IDA | Component |
| GO:0007010 | cytoskeleton organization | - | IEA | Process |
| GO:0007159 | leukocyte cell-cell adhesion | 12082081. | IEP | Process |
| GO:0008360 | regulation of cell shape | 24862762. | IMP | Process |
| GO:0008361 | regulation of cell size | 24862762. | IMP | Process |
| GO:0008361 | regulation of cell size | 22467863. | IMP | Process |
| GO:0009986 | cell surface | 15922359. | IDA | Component |
| GO:0010628 | positive regulation of gene expression | 23264465. | IGI | Process |
| GO:0016323 | basolateral plasma membrane | - | IEA | Component |
| GO:0016324 | apical plasma membrane | 24862762. | IDA | Component |
| GO:0019899 | enzyme binding | 15922359. | IPI | Function |
| GO:0019901 | protein kinase binding | 19255442. | IPI | Function |
| GO:0022612 | gland morphogenesis | 24862762. | IMP | Process |
| GO:0022614 | membrane to membrane docking | 12082081. | IEP | Process |
| GO:0030175 | filopodium | 12082081.24065547. | IDA | Component |
| GO:0031143 | pseudopodium | 24065547. | IDA | Component |
| GO:0031528 | microvillus membrane | - | IEA | Component |
| GO:0031982 | vesicle | 19190083. | HDA | Component |
| GO:0035722 | interleukin-12-mediated signaling pathway | - | TAS | Process |
| GO:0042098 | T cell proliferation | 27405666. | IDA | Process |
| GO:0045177 | apical part of cell | 12082081. | IDA | Component |
| GO:0045198 | establishment of epithelial cell apical/basal polarity | 24862762. | IMP | Process |
| GO:0048471 | perinuclear region of cytoplasm | 24862762. | IDA | Component |
| GO:0050839 | cell adhesion molecule binding | 12082081. | IPI | Function |
| GO:0050900 | leukocyte migration | 12082081. | IEP | Process |
| GO:0061028 | establishment of endothelial barrier | 23264465. | IGI | Process |
| GO:0070062 | extracellular exosome | 12519789.19056867.19199708.20458337.21362503.23533145. | HDA | Component |
| GO:0070489 | T cell aggregation | 27405666. | IDA | Process |
| GO:0071394 | cellular response to testosterone stimulus | 24065547. | IDA | Process |
| GO:0071437 | invadopodium | 25486435. | IDA | Component |
| GO:0071803 | positive regulation of podosome assembly | - | IEA | Process |
| GO:0071944 | cell periphery | 22291017. | IDA | Component |
| GO:0072562 | blood microparticle | 22516433. | HDA | Component |
| GO:0072678 | T cell migration | 27405666. | IDA | Process |
| GO:1902115 | regulation of organelle assembly | 21148287. | IGI | Process |
| GO:1902966 | positive regulation of protein localization to early endosome | 21148287. | IGI | Process |
| GO:1903364 | positive regulation of cellular protein catabolic process | 21148287. | IGI | Process |
| GO:2000401 | regulation of lymphocyte migration | 19255442. | IMP | Process |
| GO:2000643 | positive regulation of early endosome to late endosome transport | 21148287. | IGI | Process |
| Cancer | Chr | Position | Mutation Type | dbSNP | Protein-change | Allele Freq | RBD |
|---|---|---|---|---|---|---|---|
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CHOL | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| DLBC | |||||||
| DLBC | |||||||
| ESCA | |||||||
| ESCA | |||||||
| ESCA | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRP | |||||||
| LGG | |||||||
| LGG | |||||||
| LIHC | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| PCPG | |||||||
| PRAD | |||||||
| READ | |||||||
| SARC | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCS |
| Cancer | Type | Freq | Q-value |
|---|---|---|---|
| LGG | |||
| PRAD | |||
| THCA |
| Cancer | P-value | Q-value |
|---|---|---|
| KIRC | ||
| STAD | ||
| SARC | ||
| MESO | ||
| ACC | ||
| LUSC | ||
| KIRP | ||
| PAAD | ||
| LAML | ||
| GBM | ||
| LIHC | ||
| UVM |