
| Transcript ID | Name | Length | RefSeq ID | Protein ID | Length | RefSeq ID | UniportKB ID |
|---|---|---|---|---|---|---|---|
| ENST00000476482 | P4HB-210 | 765 | - | - | - (aa) | - | - |
| ENST00000576380 | P4HB-222 | 943 | - | ENSP00000460285 | 200 (aa) | - | I3L398 |
| ENST00000477607 | P4HB-211 | 1110 | - | - | - (aa) | - | - |
| ENST00000478034 | P4HB-212 | 811 | - | - | - (aa) | - | - |
| ENST00000570907 | P4HB-214 | 353 | - | ENSP00000461362 | 117 (aa) | - | I3L4M2 |
| ENST00000415593 | P4HB-202 | 1669 | - | ENSP00000388117 | 274 (aa) | - | H0Y3Z3 |
| ENST00000576541 | P4HB-224 | 571 | - | ENSP00000460741 | 188 (aa) | - | I3L3U6 |
| ENST00000439918 | P4HB-203 | 1502 | - | ENSP00000388374 | 464 (aa) | - | H7BZ94 |
| ENST00000571617 | P4HB-216 | 505 | - | ENSP00000458300 | 148 (aa) | - | I3L0S0 |
| ENST00000576052 | P4HB-221 | 562 | - | ENSP00000461791 | 166 (aa) | - | I3NI03 |
| ENST00000576390 | P4HB-223 | 439 | - | ENSP00000461718 | 106 (aa) | - | I3L514 |
| ENST00000484286 | P4HB-213 | 759 | - | - | - (aa) | - | - |
| ENST00000574914 | P4HB-219 | 571 | - | ENSP00000460612 | 156 (aa) | - | I3L3P5 |
| ENST00000575069 | P4HB-220 | 2453 | - | ENSP00000459211 | 54 (aa) | - | I3L1Y5 |
| ENST00000573778 | P4HB-217 | 688 | - | ENSP00000460088 | 208 (aa) | - | I3L312 |
| ENST00000466567 | P4HB-204 | 872 | - | - | - (aa) | - | - |
| ENST00000574007 | P4HB-218 | 1053 | - | - | - (aa) | - | - |
| ENST00000471535 | P4HB-206 | 579 | - | - | - (aa) | - | - |
| ENST00000467086 | P4HB-205 | 785 | - | - | - (aa) | - | - |
| ENST00000472244 | P4HB-207 | 858 | - | - | - (aa) | - | - |
| ENST00000571507 | P4HB-215 | 493 | - | - | - (aa) | - | - |
| ENST00000331483 | P4HB-201 | 2603 | XM_024450777 | ENSP00000327801 | 508 (aa) | XP_024306545 | P07237 |
| ENST00000473021 | P4HB-208 | 914 | - | - | - (aa) | - | - |
| ENST00000474712 | P4HB-209 | 390 | - | - | - (aa) | - | - |
| Pathway ID | Pathway Name | Source |
|---|---|---|
| hsa04141 | Protein processing in endoplasmic reticulum | KEGG |
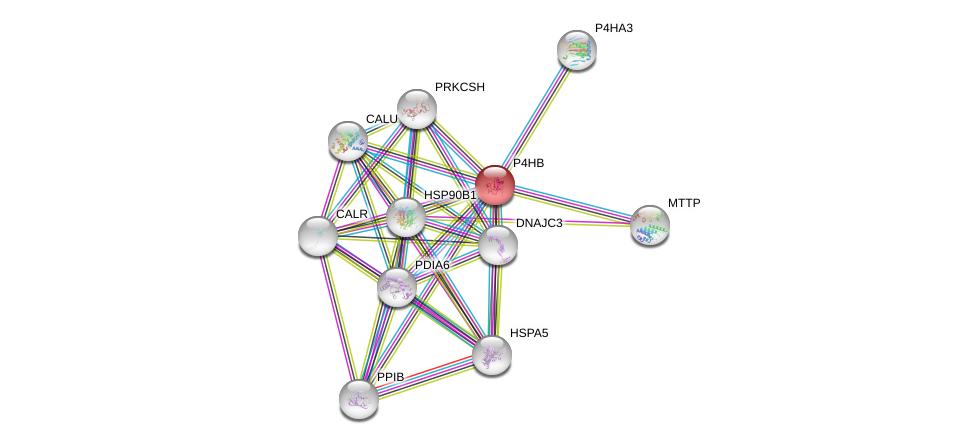
| Ensembl ID | Gene Symbol | Coverage | Identiy | Ortholog | Gene Symbol | Coverage | Identiy | Species |
|---|---|---|---|---|---|---|---|---|
| ENSG00000185624 | P4HB | 97 | 94.534 | ENSMUSG00000025130 | P4hb | 90 | 94.989 | Mus_musculus |
| Go ID | Go_term | PubmedID | Evidence | Category |
|---|---|---|---|---|
| GO:0003723 | RNA binding | 22658674.22681889. | HDA | Function |
| GO:0003756 | protein disulfide isomerase activity | 15720785. | EXP | Function |
| GO:0003756 | protein disulfide isomerase activity | 21873635. | IBA | Function |
| GO:0003756 | protein disulfide isomerase activity | 15225124. | IDA | Function |
| GO:0004656 | procollagen-proline 4-dioxygenase activity | 7753822. | IDA | Function |
| GO:0005178 | integrin binding | 21670307. | IPI | Function |
| GO:0005515 | protein binding | 12095988.17055437.19942855.20802462.21057456.21670307.23475612.23861867.25108285.26224785. | IPI | Function |
| GO:0005576 | extracellular region | 14718574. | NAS | Component |
| GO:0005783 | endoplasmic reticulum | 21873635. | IBA | Component |
| GO:0005783 | endoplasmic reticulum | 12095988.23152784.23475612. | IDA | Component |
| GO:0005788 | endoplasmic reticulum lumen | - | TAS | Component |
| GO:0005793 | endoplasmic reticulum-Golgi intermediate compartment | 15308636. | IDA | Component |
| GO:0005925 | focal adhesion | 21423176. | HDA | Component |
| GO:0006457 | protein folding | 21873635. | IBA | Process |
| GO:0009897 | external side of plasma membrane | 21873635. | IBA | Component |
| GO:0009897 | external side of plasma membrane | 21670307. | IDA | Component |
| GO:0015037 | peptide disulfide oxidoreductase activity | 16677074. | IDA | Function |
| GO:0016222 | procollagen-proline 4-dioxygenase complex | 7753822. | IDA | Component |
| GO:0018401 | peptidyl-proline hydroxylation to 4-hydroxy-L-proline | 7753822. | IDA | Process |
| GO:0019899 | enzyme binding | - | IEA | Function |
| GO:0034378 | chylomicron assembly | - | TAS | Process |
| GO:0034379 | very-low-density lipoprotein particle assembly | - | TAS | Process |
| GO:0034663 | endoplasmic reticulum chaperone complex | - | IEA | Component |
| GO:0034976 | response to endoplasmic reticulum stress | 21873635. | IBA | Process |
| GO:0034976 | response to endoplasmic reticulum stress | 12095988. | IMP | Process |
| GO:0035722 | interleukin-12-mediated signaling pathway | - | TAS | Process |
| GO:0038155 | interleukin-23-mediated signaling pathway | - | TAS | Process |
| GO:0042470 | melanosome | - | IEA | Component |
| GO:0043687 | post-translational protein modification | - | TAS | Process |
| GO:0044267 | cellular protein metabolic process | - | TAS | Process |
| GO:0045454 | cell redox homeostasis | - | IEA | Process |
| GO:0046598 | positive regulation of viral entry into host cell | 21670307. | IMP | Process |
| GO:0046982 | protein heterodimerization activity | 23475612. | IDA | Function |
| GO:0055114 | oxidation-reduction process | - | IEA | Process |
| GO:0070062 | extracellular exosome | 19199708. | HDA | Component |
| GO:0071456 | cellular response to hypoxia | 12095988. | IMP | Process |
| GO:0098761 | cellular response to interleukin-7 | - | IEA | Process |
| GO:1902175 | regulation of oxidative stress-induced intrinsic apoptotic signaling pathway | 12095988. | IMP | Process |
| PID | Title | Article | Time | Author | Doi |
|---|---|---|---|---|---|
| 29904245 |
| Cancer | Chr | Position | Mutation Type | dbSNP | Protein-change | Allele Freq | RBD |
|---|---|---|---|---|---|---|---|
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| CESC | |||||||
| CESC | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| ESCA | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| KICH | |||||||
| LGG | |||||||
| LIHC | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| PAAD | |||||||
| PRAD | |||||||
| READ | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| THCA | |||||||
| THYM | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC |
| Cancer | Type | Freq | Q-value |
|---|---|---|---|
| BLCA | |||
| CESC | |||
| KIRP | |||
| LGG | |||
| SARC | |||
| SKCM | |||
| THCA | |||
| UVM |
| Cancer | P-value | Q-value |
|---|---|---|
| KIRC | ||
| ACC | ||
| PRAD | ||
| KIRP | ||
| COAD | ||
| BLCA | ||
| CESC | ||
| KICH | ||
| UCEC | ||
| GBM | ||
| THCA | ||
| UVM | ||
| OV |