
| Protein ID | Domain | Pfam ID | E-value | Domain number | Total number |
|---|---|---|---|---|---|
| ENSP00000371994 | RNase_Zc3h12a | PF11977.8 | 1e-50 | 1 | 1 |
| Transcript ID | Name | Length | RefSeq ID | Protein ID | Length | RefSeq ID | UniportKB ID |
|---|---|---|---|---|---|---|---|
| ENST00000382554 | NYNRIN-201 | 7857 | - | ENSP00000371994 | 1898 (aa) | - | Q9P2P1 |
| ENST00000554505 | NYNRIN-202 | 425 | - | - | - (aa) | - | - |
| ensgID | SNP | Chromosome | Position | SNP-risk | Trait | PubmedID | 95% CI | Or or BEAT | EFO ID |
|---|---|---|---|---|---|---|---|---|---|
| ENSG00000205978 | rs8017377 | 14 | 24414681 | A | LDL cholesterol | 24097068 | [NR] unit increase | 0.03 | EFO_0004611 |
| ENSG00000205978 | rs11621792 | 14 | 24402720 | T | LDL cholesterol | 25961943 | [0.023-0.051] s.d. increase | 0.037 | EFO_0004611 |
| ENSG00000205978 | rs8017377 | 14 | 24414681 | T | LDL cholesterol | 20686565 | [0.8-1.54] mg/dL increase | 1.17 | EFO_0004611 |
| ENSG00000205978 | rs8017377 | 14 | 24414681 | A | LDL cholesterol levels | 28334899 | [0.023-0.038] unit increase (EA Beta value) | 0.0303 | EFO_0004611 |
| ENSG00000205978 | rs11621792 | 14 | 24402720 | C | Low density lipoprotein cholesterol levels | 29507422 | unit decrease | 0.033 | EFO_0004611 |
| ENSG00000205978 | rs11621792 | 14 | 24402720 | C | Low density lipoprotein cholesterol levels | 29507422 | unit decrease | 0.033 | EFO_0004611 |
| ENSG00000205978 | rs2332328 | 14 | 24413852 | C | Low density lipoprotein cholesterol levels | 29507422 | unit decrease | 0.036 | EFO_0004611 |
| ENSG00000205978 | rs2332328 | 14 | 24413852 | C | Low density lipoprotein cholesterol levels | 29507422 | unit decrease | 0.035 | EFO_0004611 |
| ENSG00000205978 | rs8017377 | 14 | 24414681 | G | Low density lipoprotein cholesterol levels | 29507422 | unit decrease | 0.036 | EFO_0004611 |
| ENSG00000205978 | rs8017377 | 14 | 24414681 | G | Low density lipoprotein cholesterol levels | 29507422 | unit decrease | 0.035 | EFO_0004611 |
| ENSG00000205978 | rs11621792 | 14 | 24402720 | C | Total cholesterol levels | 29507422 | unit decrease | 0.03 | EFO_0004574 |
| ENSG00000205978 | rs11621792 | 14 | 24402720 | C | Total cholesterol levels | 29507422 | unit decrease | 0.031 | EFO_0004574 |
| ENSG00000205978 | rs2332328 | 14 | 24413852 | C | Total cholesterol levels | 29507422 | unit decrease | 0.031 | EFO_0004574 |
| ENSG00000205978 | rs2332328 | 14 | 24413852 | C | Total cholesterol levels | 29507422 | unit decrease | 0.031 | EFO_0004574 |
| ENSG00000205978 | rs8017377 | 14 | 24414681 | G | Total cholesterol levels | 29507422 | unit decrease | 0.031 | EFO_0004574 |
| ENSG00000205978 | rs8017377 | 14 | 24414681 | G | Total cholesterol levels | 29507422 | unit decrease | 0.031 | EFO_0004574 |
| ENSG00000205978 | rs6573778 | 14 | 24403003 | T | LDL cholesterol x physical activity interaction (2df test) | 30670697 | EFO_0004611|EFO_0003940 | ||
| ENSG00000205978 | rs7160316 | 14 | 24401845 | ? | Breast cancer specific mortality in estrogen receptor positive breast cancer | 30787463 | [0.50-0.75] | 0.61414945 | EFO_1000649|EFO_0004352 |
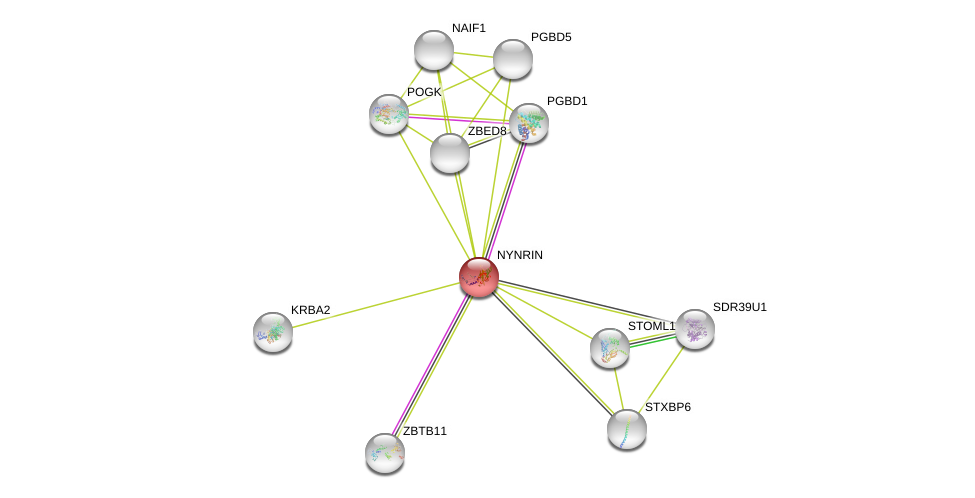
| Ensembl ID | Gene Symbol | Coverage | Identiy | Ortholog | Gene Symbol | Coverage | Identiy | Species |
|---|---|---|---|---|---|---|---|---|
| ENSG00000205978 | NYNRIN | 100 | 84.257 | ENSAMEG00000015934 | NYNRIN | 100 | 84.310 | Ailuropoda_melanoleuca |
| ENSG00000205978 | NYNRIN | 100 | 93.911 | ENSANAG00000036690 | NYNRIN | 100 | 93.911 | Aotus_nancymaae |
| ENSG00000205978 | NYNRIN | 100 | 82.730 | ENSBTAG00000007149 | NYNRIN | 100 | 83.045 | Bos_taurus |
| ENSG00000205978 | NYNRIN | 100 | 95.785 | ENSCJAG00000006385 | NYNRIN | 100 | 95.785 | Callithrix_jacchus |
| ENSG00000205978 | NYNRIN | 100 | 85.774 | ENSCAFG00000012390 | NYNRIN | 100 | 85.722 | Canis_familiaris |
| ENSG00000205978 | NYNRIN | 100 | 83.994 | ENSCAFG00020024302 | NYNRIN | 100 | 84.463 | Canis_lupus_dingo |
| ENSG00000205978 | NYNRIN | 100 | 81.618 | ENSCHIG00000017197 | NYNRIN | 100 | 82.143 | Capra_hircus |
| ENSG00000205978 | NYNRIN | 100 | 88.462 | ENSTSYG00000036783 | NYNRIN | 100 | 88.409 | Carlito_syrichta |
| ENSG00000205978 | NYNRIN | 100 | 82.838 | ENSCPOG00000014420 | NYNRIN | 100 | 82.787 | Cavia_porcellus |
| ENSG00000205978 | NYNRIN | 100 | 95.798 | ENSCCAG00000032076 | NYNRIN | 100 | 95.746 | Cebus_capucinus |
| ENSG00000205978 | NYNRIN | 100 | 95.577 | ENSCATG00000034653 | NYNRIN | 100 | 95.577 | Cercocebus_atys |
| ENSG00000205978 | NYNRIN | 100 | 84.081 | ENSCLAG00000009622 | NYNRIN | 100 | 84.029 | Chinchilla_lanigera |
| ENSG00000205978 | NYNRIN | 100 | 97.682 | ENSCSAG00000015855 | NYNRIN | 100 | 97.682 | Chlorocebus_sabaeus |
| ENSG00000205978 | NYNRIN | 100 | 97.418 | ENSCANG00000033188 | NYNRIN | 100 | 97.418 | Colobus_angolensis_palliatus |
| ENSG00000205978 | NYNRIN | 99 | 76.483 | ENSCGRG00001008471 | Nynrin | 99 | 76.931 | Cricetulus_griseus_chok1gshd |
| ENSG00000205978 | NYNRIN | 99 | 76.483 | ENSCGRG00000014985 | Nynrin | 99 | 76.931 | Cricetulus_griseus_crigri |
| ENSG00000205978 | NYNRIN | 100 | 80.733 | ENSDNOG00000049474 | NYNRIN | 100 | 80.733 | Dasypus_novemcinctus |
| ENSG00000205978 | NYNRIN | 100 | 85.825 | ENSEASG00005012250 | NYNRIN | 100 | 86.350 | Equus_asinus_asinus |
| ENSG00000205978 | NYNRIN | 100 | 85.913 | ENSECAG00000016595 | NYNRIN | 100 | 86.436 | Equus_caballus |
| ENSG00000205978 | NYNRIN | 100 | 87.434 | ENSFCAG00000022114 | NYNRIN | 100 | 87.007 | Felis_catus |
| ENSG00000205978 | NYNRIN | 94 | 94.958 | ENSFDAG00000013425 | NYNRIN | 100 | 94.958 | Fukomys_damarensis |
| ENSG00000205978 | NYNRIN | 100 | 99.210 | ENSGGOG00000016176 | NYNRIN | 100 | 99.210 | Gorilla_gorilla |
| ENSG00000205978 | NYNRIN | 98 | 84.787 | ENSHGLG00000002148 | NYNRIN | 99 | 84.947 | Heterocephalus_glaber_female |
| ENSG00000205978 | NYNRIN | 96 | 84.573 | ENSHGLG00100010082 | NYNRIN | 100 | 84.628 | Heterocephalus_glaber_male |
| ENSG00000205978 | NYNRIN | 100 | 86.660 | ENSSTOG00000024004 | NYNRIN | 100 | 86.660 | Ictidomys_tridecemlineatus |
| ENSG00000205978 | NYNRIN | 100 | 80.674 | ENSJJAG00000022144 | Nynrin | 100 | 80.305 | Jaculus_jaculus |
| ENSG00000205978 | NYNRIN | 100 | 85.759 | ENSLAFG00000014582 | NYNRIN | 100 | 85.550 | Loxodonta_africana |
| ENSG00000205978 | NYNRIN | 100 | 97.418 | ENSMFAG00000042133 | NYNRIN | 100 | 97.418 | Macaca_fascicularis |
| ENSG00000205978 | NYNRIN | 100 | 97.313 | ENSMMUG00000009513 | NYNRIN | 100 | 97.313 | Macaca_mulatta |
| ENSG00000205978 | NYNRIN | 100 | 97.313 | ENSMNEG00000032900 | NYNRIN | 100 | 97.313 | Macaca_nemestrina |
| ENSG00000205978 | NYNRIN | 100 | 97.524 | ENSMLEG00000043583 | NYNRIN | 100 | 97.524 | Mandrillus_leucophaeus |
| ENSG00000205978 | NYNRIN | 99 | 74.855 | ENSMAUG00000008402 | Nynrin | 99 | 74.698 | Mesocricetus_auratus |
| ENSG00000205978 | NYNRIN | 100 | 85.333 | ENSMICG00000031725 | NYNRIN | 100 | 84.652 | Microcebus_murinus |
| ENSG00000205978 | NYNRIN | 100 | 74.092 | ENSMOCG00000008482 | Nynrin | 100 | 74.092 | Microtus_ochrogaster |
| ENSG00000205978 | NYNRIN | 75 | 55.401 | ENSMODG00000002972 | - | 82 | 55.201 | Monodelphis_domestica |
| ENSG00000205978 | NYNRIN | 99 | 75.000 | MGP_CAROLIEiJ_G0019322 | Nynrin | 99 | 75.420 | Mus_caroli |
| ENSG00000205978 | NYNRIN | 99 | 75.342 | ENSMUSG00000075592 | Nynrin | 99 | 75.553 | Mus_musculus |
| ENSG00000205978 | NYNRIN | 99 | 75.276 | MGP_PahariEiJ_G0030339 | Nynrin | 99 | 75.066 | Mus_pahari |
| ENSG00000205978 | NYNRIN | 99 | 75.158 | MGP_SPRETEiJ_G0020211 | Nynrin | 99 | 75.578 | Mus_spretus |
| ENSG00000205978 | NYNRIN | 100 | 85.055 | ENSMPUG00000005656 | NYNRIN | 100 | 84.688 | Mustela_putorius_furo |
| ENSG00000205978 | NYNRIN | 100 | 81.437 | ENSMLUG00000011291 | NYNRIN | 100 | 81.542 | Myotis_lucifugus |
| ENSG00000205978 | NYNRIN | 100 | 80.787 | ENSNGAG00000013937 | Nynrin | 100 | 80.525 | Nannospalax_galili |
| ENSG00000205978 | NYNRIN | 100 | 97.841 | ENSNLEG00000015511 | NYNRIN | 100 | 97.841 | Nomascus_leucogenys |
| ENSG00000205978 | NYNRIN | 100 | 82.684 | ENSODEG00000001595 | NYNRIN | 100 | 82.684 | Octodon_degus |
| ENSG00000205978 | NYNRIN | 100 | 82.755 | ENSOCUG00000015163 | NYNRIN | 100 | 82.755 | Oryctolagus_cuniculus |
| ENSG00000205978 | NYNRIN | 100 | 87.710 | ENSOGAG00000024360 | NYNRIN | 100 | 88.550 | Otolemur_garnettii |
| ENSG00000205978 | NYNRIN | 100 | 80.701 | ENSOARG00000019074 | NYNRIN | 100 | 81.276 | Ovis_aries |
| ENSG00000205978 | NYNRIN | 100 | 99.368 | ENSPPAG00000028719 | NYNRIN | 100 | 99.368 | Pan_paniscus |
| ENSG00000205978 | NYNRIN | 100 | 87.382 | ENSPPRG00000004961 | NYNRIN | 100 | 86.954 | Panthera_pardus |
| ENSG00000205978 | NYNRIN | 99 | 87.114 | ENSPTIG00000016800 | NYNRIN | 99 | 86.681 | Panthera_tigris_altaica |
| ENSG00000205978 | NYNRIN | 100 | 99.262 | ENSPTRG00000006219 | NYNRIN | 100 | 99.262 | Pan_troglodytes |
| ENSG00000205978 | NYNRIN | 100 | 97.102 | ENSPANG00000018562 | NYNRIN | 100 | 97.102 | Papio_anubis |
| ENSG00000205978 | NYNRIN | 99 | 73.762 | ENSPEMG00000018953 | Nynrin | 100 | 74.619 | Peromyscus_maniculatus_bairdii |
| ENSG00000205978 | NYNRIN | 100 | 98.736 | ENSPPYG00000005705 | NYNRIN | 100 | 98.736 | Pongo_abelii |
| ENSG00000205978 | NYNRIN | 100 | 86.796 | ENSPCOG00000027737 | NYNRIN | 100 | 86.639 | Propithecus_coquereli |
| ENSG00000205978 | NYNRIN | 100 | 75.669 | ENSRNOG00000048431 | Nynrin | 100 | 76.193 | Rattus_norvegicus |
| ENSG00000205978 | NYNRIN | 100 | 97.471 | ENSRBIG00000030244 | NYNRIN | 100 | 97.471 | Rhinopithecus_bieti |
| ENSG00000205978 | NYNRIN | 100 | 97.418 | ENSRROG00000037811 | NYNRIN | 100 | 97.418 | Rhinopithecus_roxellana |
| ENSG00000205978 | NYNRIN | 100 | 95.089 | ENSSBOG00000023644 | NYNRIN | 100 | 95.089 | Saimiri_boliviensis_boliviensis |
| ENSG00000205978 | NYNRIN | 88 | 55.029 | ENSSHAG00000007338 | - | 97 | 54.947 | Sarcophilus_harrisii |
| ENSG00000205978 | NYNRIN | 85 | 84.677 | ENSSSCG00000032473 | NYNRIN | 100 | 84.738 | Sus_scrofa |
| ENSG00000205978 | NYNRIN | 100 | 85.212 | ENSUAMG00000007019 | NYNRIN | 100 | 85.265 | Ursus_americanus |
| ENSG00000205978 | NYNRIN | 98 | 80.021 | ENSUMAG00000022954 | NYNRIN | 99 | 80.075 | Ursus_maritimus |
| ENSG00000205978 | NYNRIN | 97 | 91.159 | ENSVVUG00000029210 | NYNRIN | 100 | 91.159 | Vulpes_vulpes |
| Go ID | Go_term | PubmedID | Evidence | Category |
|---|---|---|---|---|
| GO:0003674 | molecular_function | - | ND | Function |
| GO:0003676 | nucleic acid binding | - | IEA | Function |
| GO:0005575 | cellular_component | - | ND | Component |
| GO:0008150 | biological_process | - | ND | Process |
| GO:0015074 | DNA integration | - | IEA | Process |
| GO:0016021 | integral component of membrane | - | IEA | Component |
| Cancer | Chr | Position | Mutation Type | dbSNP | Protein-change | Allele Freq | RBD |
|---|---|---|---|---|---|---|---|
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BLCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| BRCA | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CESC | |||||||
| CHOL | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| COAD | |||||||
| DLBC | |||||||
| ESCA | |||||||
| ESCA | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| GBM | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| HNSC | |||||||
| KICH | |||||||
| KICH | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRC | |||||||
| KIRP | |||||||
| KIRP | |||||||
| KIRP | |||||||
| LGG | |||||||
| LGG | |||||||
| LGG | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LIHC | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUAD | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| LUSC | |||||||
| MESO | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| OV | |||||||
| PAAD | |||||||
| PAAD | |||||||
| PAAD | |||||||
| PAAD | |||||||
| PAAD | |||||||
| PAAD | |||||||
| PAAD | |||||||
| PAAD | |||||||
| PCPG | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| PRAD | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| READ | |||||||
| SARC | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| SKCM | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| STAD | |||||||
| TGCT | |||||||
| TGCT | |||||||
| THCA | |||||||
| THCA | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCEC | |||||||
| UCS | |||||||
| UCS |
| Cancer | Type | Freq | Q-value |
|---|---|---|---|
| ACC | |||
| CHOL | |||
| HNSC | |||
| KIRP | |||
| LUSC | |||
| PAAD |
| Cancer | P-value | Q-value |
|---|---|---|
| KIRC | ||
| STAD | ||
| HNSC | ||
| SKCM | ||
| LAML | ||
| UCEC | ||
| UVM |